1. 引言
根据世界卫生国际组织国际癌症研究机构(GLOBOCAN)最新的统计显示,乳腺癌已经成为世界上女性群体中最常见的癌症之一。2020年新增乳腺癌病例约200万例,占所有癌症病例的11.7%,但是乳腺癌的死亡率仅占6.9% [1] 。乳腺癌的早发现和早治疗,能够最大限度地减少诊断延误的时间,这对降低乳腺癌的致死率至关重要。
乳腺癌筛查主要是通过超声筛查和钼靶筛查,但钼靶是基于X线摄影的对乳腺内的病灶进行检查,X光是根据不同乳腺组织的密度差异进行诊断的 [2] 。有报告显示,因为“白色”乳腺组织与乳房X光检查中显示的“白色”乳腺癌组织之间缺乏一定的对比 [3] ,而超声可检测出乳房X线照片中难以发现的乳腺癌 [4] ,可提升乳腺组织致密者中的乳腺癌检测的准确率 [5] ,X光在诊断这类患者方面具有一定的局限性 [6] 。在乳腺组织密度较小的患者中,超声图像检测也要优于X光 [7] 。此外超声检测相对于X光检测无辐射,而且超声检测是实时动态的,以便于观察图像,确定病情。
近年来,计算机辅助诊断技术(Computer Aided Diagnosis, CAD) [8] 在医学图像领域取得了飞速发展。他在提高诊断准确性、减少漏诊、提高工作效率等方面发挥了重大作用。
机器学习方法已被用于医学领域的检测和诊断,其中支持向量机是最常用的机器学习算法之一,通过提取乳房X线图像ROI区域的灰度共生矩阵(GLCM)纹理特征进行分类,支持向量机的准确率可达94% [9] 。通过提取乳腺超声图像的一阶统计纹理特征进行分类,支持向量机分类的总体准确率为86.6% [10] 。在机器学习中另一个常用的分类器是决策树,它也是一种基本的分类方法。在某一心脏病识别任务中,分别使用了三种算法:Decision Tree、Bagging with Decision Tree和Bagging with Naive Bayes,三种模型的准确率分别为78.93%、79.53%和82.50% [11] 。梯度提升算法是近几年预测模型中最强大的机器学习算法。文献 [12] 中使用梯度提升决策树–多参数优化器(Gradient boosting decision tree - Multi-verse optimizer, GBDT-MVO)模型证明了它在乳腺癌诊断中具有更高的精度和较低的方差。
深度学习的相关算法在医学图像处理任务上也发挥了重大作用。FCN [13] 是图像分割的里程碑,具有全监督的全卷积神经网络(FCNN)在肝脏图像的血管分割指数DICE中达到0.79 [14] 。U-Net [15] 网络的提出是对医学图像语义分割的重大贡献,同时也激发了大量研究人员对U型分割网络的思考。受U-Net及其变体的启发,所提出的NAS-UNET由堆叠在U-Type主干上相同数量的DownSC和UpSC组成,其结果在三种不同类型的医学图像数据集上表现出良好的分割性能 [16] 。有很多新的分割网络都是在U-Net结构的基础上改进的,但正如nnU-Net [17] 文中提到的那样,这些被称为SOTA的网络结构大都是基于U-Net结构改进而来的,如nnU-Net,一些网络结构如SOTA都是基于U-Net结构而针对特定数据集进行特定的调整参数,而不是本质上的改进。因此,U-Net在医学图像分割邻域中仍具有不可动摇的地位。
在本文中,我们基于U-Net框架对整个超声图像进行分割,并计算区域的肿瘤形状特征和纹理特征。使用四种不同的机器学习算法对这些功能进行了重组和验证。在验证结果中,选择最好的特征和分类的模型来评估测试集(图1)。

Figure 1. Breast ultrasound diagnostic model
图1. 乳腺超声诊断模型
2. 研究方法与相关理论
2.1. LightGBM [18] (Light Gradient Boosting Machine)算法
LightGBM是一种用于机器学习的快速梯度提升算法。它主要用于分类和回归任务。
LightGBM的优势在于,它可以处理大规模数据集,并且在训练过程中使用多种优化技术,使得它比其他算法 [19] [20] 更快。它还能够处理稠密数据和稀疏数据,并且具有较高的准确性。
2.2. U-Net网络
UNet是一种适用于图像分割的深度学习模型。UNet的结构非常适合处理医学图像分割任务。它的编码器负责从图像中提取有用的特征,而解码器则负责将这些特征转换为最终的分割图像。UNet的跳连接层,可帮助网络更好地学习图像中的细节。这些特点使得UNet能够在保留图像细节的同时准确地对图像进行分割。此外,UNet还具有较少的参数,这意味着它需要较少的训练数据来调整模型。这在医学图像领域中尤为重要,因为医学图像数据常常是非常有限的。总的来说,UNet的准确性、细节保留能力和较少的参数使它成为医学图像分割的理想选择。
2.3. 形状特征
根据医生的临床经验,肿瘤的形状特征是判断肿瘤是否为恶性的标准之一。因此,以下这些形状特征将被计算用于肿瘤分类。
椭圆相似率 [21] (ESR):最佳拟合椭圆 [7] 是通过直接最小二乘拟合椭圆算法获得的。如图2所示,深蓝线为肿瘤轮廓,黄线为最佳拟合椭圆。椭圆相似度是通过最佳拟合椭圆的周长与原始肿瘤轮廓周长的比率来计算的。如果原始肿瘤轮廓的形状特征规则,则与椭圆相似度的值更接近1。椭圆相似度(ESR)定义如下,Pe和Pt分别代表最佳拟合椭圆周长和肿瘤轮廓周长。
偏转角(DA):肿瘤的偏转角是肿瘤轮廓最佳拟合椭圆长轴与水平方向的偏转角。如图2所示,θ为肿瘤的偏转角。DA定义如下。
形状紧凑率(FCR):致密性可以衡量肿瘤边缘的缺损程度。良性肿瘤的形状通常比恶性肿瘤更规则,缺损程度更低。形状紧实度(FCR)定义如下,Ar代表肿瘤的面积,C代表肿瘤轮廓的周长。
横向纵向比(TLR):肿瘤的横纵比可以通过计算肿瘤的外接矩形的长高比来计算,外接矩形是通过最高点、最低点、最左点和最右点形成的矩形。如图2所示,红色虚线矩形区域为肿瘤的外接矩形。肿瘤横向纵向比(TLR)定义如下,T代表外接矩形的长度,L代表外接矩形的高度。
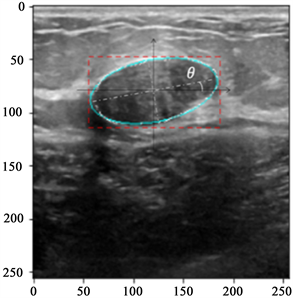
Figure 2. Different contour lines for different shape features
图2. 不同形状特征的不同轮廓线
2.4. 纹理特征
灰度共生矩阵中的保存的数据量很大,一般不直接用作区分纹理特征,而是将基于灰度共生矩阵构建一些统计量用作纹理分类特征。哈拉利克 [22] (Haralick)给出了些基于灰度共生矩阵计算的14个统计量:角二阶矩、对比度、相关性、方差、逆差矩、求和平均、求和方差、求和熵、熵、差方差、差熵、相关性1的信息度量、相关性2的信息度量、最大相关系数。在本研究中,选取13个统计量(不包括最大相关系数)作为提取的纹理特征。由于灰度共生矩阵中θ有四个方向,可以得到四组统计量,再加上四组统计量的平均值,可以得到五组统计量,共65个特征,即被用作本文提取的纹理特征。
3. 乳腺超声诊断模型的方法
3.1. 乳腺超声诊断模型概述
本文乳腺超声诊断模型如图1所示,先利用训练集训练一个U-Net分割模型,得到每个肿瘤区域的mask,根据原图与对应的mask划分出信息一起进入特征提取模块,再进行特征组筛选和分类器评估,以得到最终的最佳模型。
3.2. 图像数据集
在研究中,数据主要来自数据集DBUI (Data of Breast Ultrasound Images) [23] 。该数据集包括从600名女性患者获得的780张乳房图像,所有图像的格式均为PNG类型。这些图像分为三类:正常、良性和恶性。本此研究选取399份良性肿瘤样本和183份恶性肿瘤样本作为实验数据。每个图像都有其对应的分割掩码(图3)。
按照比例随机分为训练集和测试集,其中训练集用于训练分割模型(3折交叉验证)和特征选择及分类器选择(10折交叉验证)。在训练超声图像分割时的训练集是基于扩增的训练集数据进行训练,训练集数据还会进行特征选择和分类模型选择,然后使用测试集来对最终整体模型的结果进行评估。
3.3. 分割网络
在本研究中,分割的网络框架基于U-Net网络,参考了nnU-Net的思想,对原始数据进行了一些预处理。
对训练集使用随机旋转、水平翻转和垂直翻转等技术增强图像,在训练集上使用了3折交叉验证,损失函数结合了Dice损失和交叉熵损失。
3.4. 特征提取
特征提取流程图详见图4。
1) 形状特征提取,基于分割网络的得到的肿瘤内部区域mask去计算形状特征,分别是椭圆相似率、偏转角、边缘紧凑率、肿瘤横纵比。
2) 纹理特征提取,纹理特征从以下4个不同的区域中提取(图5)。a区域是分割得到的mask,b区域是mask膨胀后减去原始mask得到的一个环形区域,c区域是mask膨胀算法后的外矩形,d区域是整个图像。这四个区域划定规则是由肿瘤内部由内向外依次扩大范围得到的区域。因为从临床评估上来说肯定是优先关注肿瘤区域再关注逐渐关注肿瘤周围区域,这四个区域的重要性在后续实验中会进一步验证。由于这四个区域的纹理特征均来自同一张超声图像,因此这四个区域的纹理特征将作为同一样本的纹理特征。
3) 降维:从每个超声图像中可以获得264 (260 + 4)个特征。这些特征包括从四个不同区域获得的4个形状特征和4个不同区域获得的260 (65 × 4)个纹理特征。降维特征组是对具有高相关性的冗余特征进行降维得到的。在本文中,将采用LASSO算法 [24] 对特征降维进行计算。
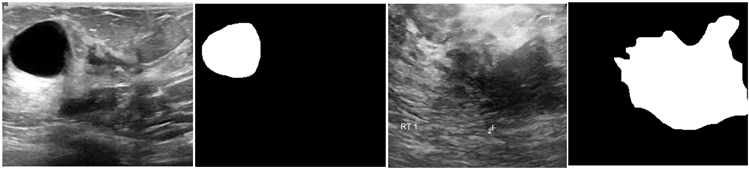
Figure 3. Benign and malignant ultrasound breast tumors and their corresponding masks
图3. 良性和恶性超声乳腺肿瘤及其相应掩码
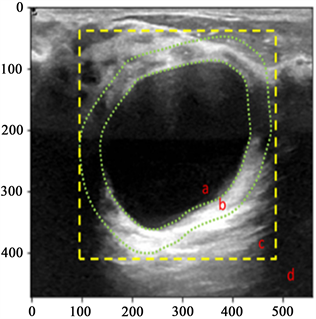
Figure 5. Different areas of breast ultrasound images
图5. 乳腺超声图像的不同区域
3.5. 特征重组
根据不同的区域和不同的类型对特征进行重新组合,得到不同的组合特征集,然后由分类器选择最佳特征集。
3.6. 分类器
在正如2.1节提到的我们在数据集上训练和测试LightGBM (Light Gradient Boosting Machine)分类器。我们还在同一数据集上训练和测试了其他 3个分类器,将它们的性能与LightGBM分类器进行比较。3种算法包括K-最近邻 [25] (K-Nearest Neighbor),支持向量机(Support Vector Machines),随机森林 [26] (Random Forest)。
3.7. 评价指标
为了更全面地评估每个模型的性能,我们使用了如下几个模型评估指标。在分割任务中,利用DSC (Dice Similarity Coefficient)、IOU (Intersection Over Union)、Recall、Precision、Global Accuracy和AUC来评估分割性能。
使用AUC、ACC、Sensitivity和Specificity来评估分类性能。
4. 实验结果分析
4.1. 分割结果
从数据集中总共选择了582张图像,并将它们分为良性和恶性两类,作为分割数据。其中随机划分482张训练集图像和100张测试集图像。为了达到更好的网络训练效果,我们通过水平翻转、垂直翻转、左旋转和右旋转来增加训练样本以得到4338个训练样本,并将它们与U-Net模型进行3折交叉验证。将性能最好的分割网络用于在测试集上进行测试。最终结果如下(表1,图6)。
从结果中我们发现,经过数据预处理后训练得到的模型的分割性能较之前提高了10%左右。DSC、IOU和Precision的最终结果分别为0.778、0.703、0.899。
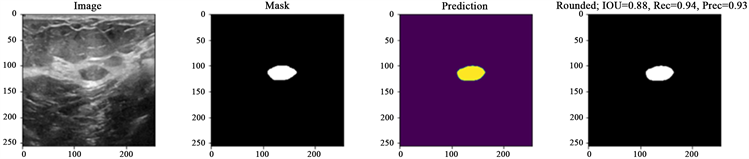 (a)
(a) (b)
(b)
Figure 6. Segmentation results for benign and malignant tumors. (a) Benign tumor segmentation; (b) Benign tumor segmentation
图6. 良性和恶性肿瘤的分割结果。(a) 良性肿瘤分割结果图示;(b) 恶性肿瘤分割结果图示
4.2. 特征选择结果
4.2.1. 特征重组
重新排列组合方式见表3,我们之前特征提取得到的5组特征,就是对应表3中的特征组NO.1-NO.5,重组规则是按照肿瘤位置区域依次往外扩大区域,再依次结合形状特征得到6组复合特征组就是对应表3中的特征组NO.6-NO.11,其中特征组NO.11是复合了全部5组特征。特征组12就是对特征组11在LASSO上降维得到的特征组(NO.12) (见表2),因此得到一共12组特征(见表3),我们并没有直接将降维特征组NO.12作为我们的最佳特征组。而是通过后续实验从这12特征组(表3)中进一步验证从而选出最佳的特征组合。
4.2.2. 实验结果
上面12个不同的特征集在4个分类器中进行了10倍交叉验证。图7显示了4个分类器上12个特征组中的ROC曲线。表4是各特征组和分类器对应的具体指标性能。对12个特征组指标可以看出,形状特征(特征NO.1)明显优于其他单组特征(特征组NO.2-NO.4),而从单从纹理特征组(NO.2-NO.4)可以看出,单独的内部纹理特征组(区域a,特征组NO.2)性能最好,环状纹理特征组(区域b,特征组NO.3)性能其次,另外两组纹理特征(区域c、d,特征组NO.3、NO.4)性能指标可以看出没有明显区分性,就复合特征组而言(NO.6-NO.11),随着复合的特征越多维度越高,对应的指标也是在提升的,但与之对应的是会有很多冗余特征,所以对应的经 LASSO 降维后的特征组(NO.12)性能优于其他任何复合组。对四种分类器的比较可以看出,LightGBM算法性能明显优于其他三种分类器,因此实验得出结论是最佳特征组为经 LASSO降维后的特征组(NO.12),最佳分类器为LightGBM。

Table 2. Characteristic coefficients screened by LASSO algorithm
表2. LASSO算法筛选的特征系数

Table 3. Feature recombination information
表3. 特征重组信息

Table 4. Performance of 12 sets of features on 4 classifiers
表4. 12组特征在4个分类器上的性能表现
 (a)
(a) 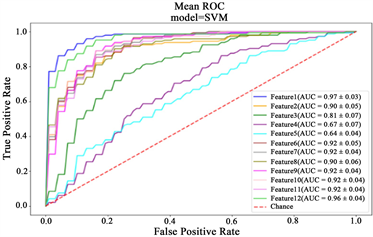 (b)
(b) 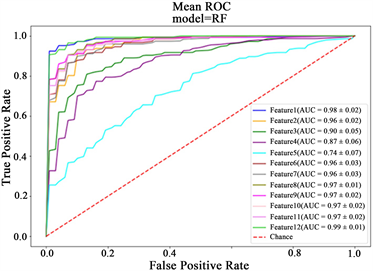 (c)
(c)  (d)
(d)
Figure 7. The average ROC curve obtained by 10-fold cross-validation of 12 sets of features on 4 classifiers. (a) KNN ROC curve; (b) SVM ROC curve; (c) RFROC curve; (d) LightGBMROC curve
图7. 12组特征在4个分类器上通过10折交叉验证得到的平均ROC曲线。(a) KNN ROC曲线;(b) SVM ROC曲线;(c) RF ROC曲线;(d) LightGBM ROC曲线
4.3. 训练和测试
我们将数据集按照7:3的比例划分为训练集和测试集,通过对训练集进行10倍交叉验证得到最佳特征组和最佳分类器(这些工作都是在训练集上完成的)。最终模型需要重新训练和测试以保证客观性。首先,从我们提取的所有特征中选择最佳特征组(降维特征组12)作为数据特征,用最好的分类器(LightGBM)用训练集数据重新训练得到模型,然后对模型用测试集进行测试,最终测试结果如表5所示。最终结果的准确度、灵敏度和特异性分别为0.966,分别为0.98、0.938。
4.4. 与深度学习相关算法对比
为了进一步验证我们所提出的模型的性能优势,我们选取了常用的几种深度学习模型进行对比。这几组深度学习初始权重都是在ImageNet上进行预训练得到的,在我们的划分的乳腺超声图像训练集上训练得到最佳参数,再在测试集上测得性能指标见表6。可以看出,我们所提出的方法性能指标明显高于深度学习各个算法。

Table 5. Performance of the model on the test set
表5. 模型在测试集上的表现

Table 6. Compare with the deep learning models
表6. 与深度学习各个模型对比
5. 讨论
在此项中,良性样本与恶性样本的比例接近2:1,存在数据不平衡的问题。解决分类任务中的数据不平衡问题主要有三种方法 [35] :1) 过采样或欠采样,通过增加少数类样本或减少多数类样本。2) 采用代价敏感型学习方法该方法根据不同类别的错误分类样本数去重新定义损失函数。3) 集成学习。该方法将集成学习方法与数据重采样技术相结合。在本文中,我们尝试按照训练集类比例给样本添加权重,结果表明增加权重后各项指标确实均有一定提升(参考表5中均衡权重组)。
与传统算法相比,深度学习可以更好地学习到样本数据的内在规则。深度学习可能需要更大的数据量以支持系统进行训练模型。然而在医学领域,很难获得大数据集,只有小样本数据集可用,因此在这本文中,我们仍然使用传统的算法(LightGBM)作为分类器并取得了不错的效果。
在医学图像领域中,前处理和后处理的效果有时会大于网络本身的变换。从不同分类器下不同特征的结果可以发现,与其他特征组相比,大部分分类器对于具有单个形状的特征组都具有较好的分类效果。内部区域和环形区域的纹理也表现出一定的分类性能,但单独不同区域的纹理特征并没有表现出优异的性能,原因可能是图像的纹理特征与设备的类型有关。从实验结果可以看出,从医学图像中提取临床病理特征对最终诊断提升是有一定意义和价值的,如果基于各种图像算法盲目地提取大量特征可能会在一定程度上对结果有一定的增强,但会大大降低临床可解释性。
6. 结语
在本文中,我们提出了一种全流程的乳腺超声诊断系统,结果表明我们所提方法准确率达到0.966,灵敏度达到0.98,特异度达到0.93。可以实现乳腺超声图像良恶更高精度的诊断,帮助医生提高超声乳腺图像诊断的效率和准确性。具有一定的研究价值和临床意义。
利益冲突声明
本文全体作者均声明不存在利益冲突。
NOTES
*通讯作者。