摘要: 本文主要从UCSC Xena数据库中已经整理好的关于TCGA数据库的乳腺癌数据中,挑选了拷贝数变异、RNA基因表达量、RNA外显子表达量三个组学方面的数据。首先,基于三个组学数据的维度远大于样本量的特征,分别对三个组学的数据进行方差阈值过滤,初步筛选过滤掉变化幅度不大的变量,再使用mRMR进行滤波式的变量选择方法,即最大化特征与分类变量之间的相关性,最小化特征之间的相关性,各自筛选得到50个变量。对于离散型的天数表型数据,采用阈值方法将其转化为0-1分类变量,最终将因变量与自变量进行合并,并划分测试集、训练集,使用svm、XGBoost、Logistic、RandomForest四种方法对结果变量进行预后预测,并采用特定的指标对这四种算法进行比较,运用在训练集上,最终得到XGBoost、Logistic两种算法的预测效果要优于svm、RandomForest。
Abstract:
In this paper, we mainly selected the three omics data of copy number variation, RNA gene expres-sion, and RNA exon expression from the breast cancer data on the TCGA database that have been collated in the UCSC Xena database. Firstly, based on the characteristics of the three omics data whose dimensions are much greater than the sample size, the variance threshold filter is performed on the three omics data, the variables with little change are filtered out initially, and then the vari-able selection method is filtered by using mRMR, that is, to maximize the correlation between the features and the categorical variables, minimize the correlation between the features, and filter 50 variables each. For the discrete number of days phenotypic data, the threshold method is used to convert it into a 0-1 categorical variable, and finally the dependent variable is merged with the in-dependent variable, and the test set and the training set are divided, and the outcome variable is predicted by svm, XGBoost, Logistic, randomForest, and the four algorithms are compared with spe-cific indicators, and the training set is applied to the training set, and finally XGBoost. The predic-tion effect of logistic algorithms is better than that of svm and RandomForest.
1. 引言
世界卫生组织于2020年发布了最新版全球癌症报告,报告中指出预计中国癌症新发病例和死亡病例的比例约占全球癌症发病和死亡的23.7%、30.2%,均高于我国人口占全球人口的比例 [1],其中,乳腺癌已成为女性癌症致死的首位原因(15.5%)。近年来,随着生物、医学等学科的发展,为疾病的预测和治疗提供了无限可能,但在现有的医疗水平下,我国癌症死亡人数比例仍高达全球癌症死亡总人数的30%。超过半数以上的癌症,发现时处于晚期,而晚期的治愈率远不如早期,如:肾细胞癌,两年内的生存期仅达13.4% [2];非小细胞肺癌患者的死亡率高达75%等。由于缺少准确的病情发展预测和未能接受及时的治疗,错过了治疗的黄金时期。预后分析模型既满足了患者对可用于规划生活的未来信息的需求,又为合理的医疗决策提供了基础,因此,进行合理、准确的预后分析是十分有必要的。近年来,针对癌症的预后分析受到国内外学者的广泛关注,而早在公元400年前希波克拉底学派就意识到环境的因素和患者的特征等都是影响预后的因素 [3]。比如,临床症状相同的癌症患者,其预后结果可能相去甚远。此外,基因差异的表达也会对患者的治愈产生显著性差异。比如处于同一时期的乳腺癌患者关于PGLYRP2、SEMA3G、PROL1及SLC7A3基因的高表达,其预后效果要优于SKA1、BIRC5、RRM2和AURKA基因高表达的患者 [4]。乳腺癌主要与基因组改变、激素破坏、代谢异常、蛋白质失效、信号通路改变以及环境因素等不同生物学方面之间的复杂相互作用有关 [5]。而随着基因组学与影像组学广泛用于肿瘤的精准诊疗,对乳腺癌组织病理图像的研究也越来越多。因此,部分基于人工特征提取和传统机器学习算法被运用到乳腺癌病理图像分类上,比如Haralick等 [6] 提出了一种经典的灰度共生矩阵(Gray-Level Co-occurrence Matrix, GLCM)来提取图像的纹理特征、Kowal等 [7] 采用自适应阈值技术和高斯混合聚类对乳腺癌组织病理学图像中的细胞核进行分割,在500幅乳腺癌病理图像上的准确率为92%~98%。近几年,随着计算机性能的不断增强,一些训练深度网络的新技术也被广泛应用到医学图像识别和分类领域。这方面的贡献包括:Araújo等 [8] 使用神经网络提取乳腺癌组织病理学图像的深层特征,并使用支持向量机进行分类,二分类准确率高达90%,四分类最高准确度为85%;Han等 [9] 在BreaKHis数据集下进行了分类实验,提出了一种基于类结构的深度卷积神经网络,得到了二分类、多分类均高达90%以上的准确率。
在本文中,我们选取三个组学的数据,采用特定的模型对乳腺癌病人进行预后预测。我们对三个组学的数据使用mRMR方法进行特征选择,分别筛选各自得到50个特征,将这些特征进行结合,得到一个具有150维的多组学数据,根据这些特征分别采用XGBoost、svm、RandomForest、Logistic四种方法进行预后预测。其中,XGBoost是一种基于GBDT梯度提升的算法,它利用添加一棵树来达到添加一个学习函数f(x)的效果,用来拟合上一次的预测残差,具有高效、灵活的特点;svm是一种超平面分割的算法,利用对偶性将问题进行转换,由于其计算量极大,因此核函数的选取也十分重要;Logistic模型本质上是一种回归模型,用于估计事件发生的可能性,因此也可用于分类问题;randomForest是一种结合多颗决策树的集成算法,由于其选择特征、样本的随机性,使得模型的泛化性良好。最后,使用模型预测精确度、AUC、ROC曲线指标,来评估方法的性能,根据这三种指标,均能得出XGBoost、Logistic二者算法的预测效果要优于svm、RandomForest。
2. 材料和方法
2.1. 数据集
本文数据集来自UCSC Xena官网关于TCGA数据库的1000多个乳腺癌病人的多组学数据,包括:拷贝数变异、RNAseq表达量、外显子三个组学数据,在进行一系列缺失值处理以及样本合并后,得到一个最终维度远大于样本数的完整数据集。
表型数据是患有乳腺癌病人的生存天数,首先将该离散型数据因子化,即存活时间大于五年的,记为1,存活时间小于五年的,记为0,得到0-1型的因变量。对于exon组学数据,由于其具有二十多万个变量,因此先在该组学数据内部进行相关性检验,与因变量间相关系数小于0.1的变量,说明二者之间的相关性不是很强,予以剔除,最后得到两万多个自变量。
其次,分别计算三个组学的自变量方差大小,剔除其中最小的10%的变量,将通过方差阈值过滤得到的三组数据,再分别采用mRMR方法进行变量特征筛选,三个组学数据的维度见表1。

Table 1. Dimensions of multi-omics dataset
表1. 三个组学数据维度
2.2. 特征提取
虽然本文数据中的选择变量很多,但是很多变量间提供了重复的信息,即变量间存在冗余的关系,这可能会使得模型变得复杂,某种程度上增加模型训练的成本。在机器学习领域,我们通过降维的方法来化解这一复杂的问题,其方法有多种,比如主成分分析、LASSO回归等;而本文采用的是mRMR算法 [10],在特征提取领域具有广泛的应用,它是一种通过最大化特征与目标变量之间的相关性的算法,利用互信息论,通过建立冗余度和相关性,来降低特征维数。本文选取的三个组学数据中,经过初步的方差过滤,仍存在变量维数较高的问题,故采用该算法分别对三组数据进行降维,筛选后各自得到1080 × 50的数据矩阵。
2.3. svm模型
svm [11] 本质上是一种二分类模型,其本质是使得特征空间上间隔最大化的分类器,即找到一个超平面,使得支持向量这一超平面之间的间隔、距离最大化,最终转化为一个凸二次规划问题求解。其算法的本质如下,利用点到超平面空间的距离,且位于超平面两端的样本点都被正确分类,调整相应参数,得到目标函数,最终转为一个带约束的目标优化函数,通过拉格朗日乘子法,再利用KKT条件将原问题转化为对偶问题,最终得到最优解。其中,在转化为最优求解问题的过程中,由于内积算法的复杂度,可采用不同的核函数来降低算法的复杂性,常见的核函数有线性核函数(linear kernel function)、多项式核函数(polynomial kernel function)、高斯核函数(gaussian kernel function)以及sigmoid核函数等。
2.4. XGBoost模型
XGBoost [12] 本质上是一种boosting类别的集成学习算法,在GBDT的基础上,从目标展开函数、拟合标签、添加正则项以及自动处理缺失值这几个方面进行一定的优化,使其在性能和效果上有一定的提升。其核心算法思想是:
1) 结合贪心算法,不断的添加树,不断地进行特征分类来生长一棵树,然后进行损失函数的计算,找到最小的损失值,分裂该树,利用添加一棵树来达到添加一个学习函数f(x)的效果,去拟合上一次的预测残差;
2) 当我们训练完样本得到m棵树,想要得到相应的预测样本的分数,就是根据这一样本的特征,在每棵树中会有一个对应的叶子节点,每个叶子几点就对应一个相应的分数;
3) 最后,将每棵树对应的分数值相加,就是该样本的预测值。
2.5. Logistic回归
Logistic回归是一种广义上的线性模型,常用于数据挖掘,疾病自动诊断,经济预测等领域。其核心思想是对线性函数作不同的变换,若y为二分类变量,这一转化将其范围转到从0到1上,再采用极大似然估计法多次迭代求解,得出最终的系数。对于普通的线性模型来说,其数据分布假设可放宽到满足正态分布族即可,使用范围更广,针对本文中的因变量满足二项分布,也是正态分布族中的一员,因此可使用该模型进行预后预测。
2.6. RandomForest随机森林
RandomForest随机森林可以看作是决策树的一种boosting集成算法,随机选取若干样本、特征,生成若干颗决策树,由这些决策树,构成一片森林。正是由于这一算法的随机性引入,使其容易避开过拟合这一问题,并且具有较好的抗噪能力;它对数据集的适应性也很强,既能处理离散型数据,也能处理连续性数据。本文详细流程图见图1。

Figure 1. The flow chart of methods proposed in the paper
图1. 本文方法流程图
2.7. 评估准则
对于二分类问题,有许多的评价准则,本文选取混淆矩阵(Confusion matrix)、对结果性能进行评估(见表2):
其中,P (Positive):代表1;N (Negative):代表0;T (True):代表预测正确;F (False):代表预测错误;ROC曲线(Receiver Operating Characteristic Curve)以及AUC (RecieverOPeration Characteristic):ROC曲线以FPR为横坐标,TPR为纵坐标,样本数量越多,ROC曲线越平滑,AUC值是ROC曲线下方的面积,越接近于1,模型效果越好,越接近真实效果。
3. 结论
特征变量过多,可能带来信息冗余,提升模型的复杂度,从而导致过拟合、计算效率过于低下等问题。本文中,在进行特征提取之前,首先各自可视化了各个组学数据的方差波动情况(见图2),发现RNAseq这一组学数据对应的方差波动较大,说明该组学数据的变量可能提供更多的潜在信息,有了这一先验,进而对各自组学数据进行特征筛选。对于最后得到的筛选数据集,再进行样本5次的平衡测试集、训练集划分,将训练集与测试集的样本比例保持在4:1,且对因变量的每一类随机抽样,保持其划分数据集前后整体类别的比例一致,更有利于模型的泛化。
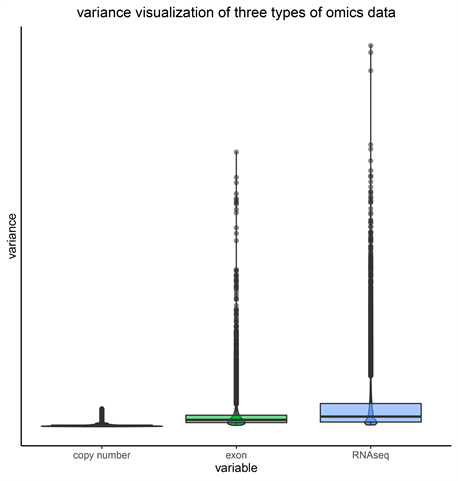
Figure 2. Variance visualization of three types of omics data
图2. 三个组学数据的变量方差可视化
在训练集完成模型学习之后,分别对划分的五次测试集进行支持向量机、XGBoost、随机森林、Logistic四种算法预后预测,利用每个模型得到的预测结果形成相应的混淆矩阵,并计算TP、TN二者所占的比例,分别得到的五个精度(见表3),从下表可看出XGBoost、Logistic这两种算法的预测效果要优于另外两种。

Table 3. The prediction accuracy of the four algorithms on each of the five test datasets
表3. 四种算法分别在五个测试集上的预测精度
观察下图(见图3),蓝色曲线代表XGBoost算法,紫色曲线代表SVM算法,红色曲线代表Logistic回归,黄色曲线代表RandomForest算法,虚线y = x代表随机猜测,可以看出,SVM、RandomForest算法下的面积即AUC的值相对于其他两种算法来说较小,XGBoost、Logistic算法下的AUC值较大,也从另一方面说明本文中,后两者的算法优于前两者的算法。
4. 总结
本文将三种组学类型的数据通过mRMR算法进行降维之后,结合在一起形成一个新的数据集,将得到的150个变量作为新的特征向量集。然后基于这一新的多组学数据分别进行四种机器学习方法的训练,将最后得到的模型用于测试集上,对四种方法进行精度比较,得到XGBoost、svm、Logistic、RandomForest四种算法的精度分别为0.8、0.68、0.792、0.777。