1. 引言
肌酐水解酶(Creatinine hydrolase, EC:3.5.2.10)是一种存在于微生物中的酶,可以可逆的催化肌酐水解为肌酸,在分解肌酐的体系中位于首位。主要作用是使含有此种代谢通路的微生物可以以肌酐为氮源,以进行生命活动。目前肌酐水解酶主要应用于血清肌酐、尿肌酐浓度的测定中,本文总结了其理化性质、催化机理及应用等。
2. 肌酐水解酶的发现、理化性质及其分离纯化方法
2.1. 肌酐水解酶的发现
肌酐水解酶最早在1950年被发现,和它同时被发现的还有其下游的肌酸水解酶 [1];1974年,肌酐水解酶在节杆菌中被分离纯化出来,对其性质也有了初步了解 [2] [3];此后1979年,Rikitake等在恶臭假单胞菌中分离纯化出一种肌酐水解酶,并对其性质进行了深入研究 [4]。
2.2. 肌酐水解酶的理化性质
在肌酐水解酶中,恶臭假单胞菌来源的肌酐水解酶研究比较充分,以下主要介绍恶臭假单胞菌来源肌酐水解酶的理化性质,并对其他来源的肌酐水解酶做简要介绍。恶臭假单胞菌来源的肌酐水解酶是一种酸性金属酶,等电点为4.7左右;在天然状态下一个亚基中存在两个锌离子,其中一个锌离子易被锰离子取代,取代后的肌酐水解酶酶活有所提高。肌酐水解酶为同源六聚体,每个肌酐水解酶亚基的分子量在27~29 KDa左右,由250~268个氨基酸组成,其中每两个亚基组成一个二聚体,三个二聚体再组成同源六聚体,即完整的肌酐水解酶。肌酐水解酶具有较强的稳定性:在pH 6~12的范围内较为稳定,并在pH 7~9的条件下保持较高的酶活;能耐高温,在75℃的条件下处理30 min仍能保持75%的酶活;具有较强的稳定性,对EDTA处理并不敏感,使用10 mM EDTA在pH 7.1的50 mM Tris-HCl缓冲液中30℃处理30 min,仍能保持80%~90%的酶活 [4]。肌酐水解酶可以可逆的催化肌酐水解为肌酸,此反应受反应体系温度及pH的影响,随着温度的升高、反应pH的上升,反应的平衡向生成肌酸的方向移动,反之亦然。
2.3. 肌酐水解酶的分离纯化
不同来源的肌酐水解酶具有类似的理化性质,因此其分离纯化步骤也基本相似,都使用了加热、盐析、阴离子层析和分子筛层析等方法进行分离纯化。来自Arthrobacter ureafaciens的肌酐水解酶通过55℃水浴加热、10%链霉素溶液除核酸、35%饱和度硫酸铵盐析、Sephadex G-200层析和DEAE阴离子层析5步操作得到了纯酶 [3]。来自Pseudomonas putida var. naraensis, strain C-83的肌酐水解酶使用了鱼精蛋白除杂、亲和层析、热处理、DEAE阴离子层析和Sephadex G-200五步得到了纯酶 [4]。来自Pseudomonas putida的肌酐水解酶基因被克隆至大肠杆菌BL21 (DE3)中后,在C端加入6 × HIS-tag,仅使用热处理、镍柱层析和分子筛层析3步就得到了纯酶,简化了分离步骤,并且提高了回收率 [5]。
3. 肌酐水解酶酶活测定方法、结构及催化机制
3.1. 肌酐水解酶酶活测定
肌酐水解酶酶活的测定有两种方法,其中较为常用的为使用Jaffé法的酶活测定方法,此法以肌酸为底物,在肌酐水解酶的催化作用下使肌酸转化为肌酐,肌酐在碱性条件下与苦味酸发生反应,生成橘黄色产物,在520 nm的可见波长下其浓度与吸光度成正比,因此可以通过测定肌酐的生成量来测定肌酐水解酶酶活。
另一种为以肌酐为底物,通过α-二乙酰萘酚法测定肌酸生成量来测定酶活。在肌酐溶液中加入酶液后,肌酐被酶催化为肌酸,肌酸同样可以在碱性条件下与二甲基乙二醛和α-二乙酰萘酚反应,其产物的吸光度值在540 nm的可见波长下进行测定,进而测定肌酐水解酶酶活。
3.2. 肌酐水解酶结构及催化机制
在2004年通过X射线衍射法解析了恶臭假单胞菌来源的肌酐水解酶的结构,分辨率达到了1.8 Å。确定了恶臭假单胞菌来源的肌酐水解酶含有6个相同的亚基,每个亚基由7个α螺旋和5个β链组成α/β-型结构,每两个亚基组成同源二聚体,三个同源二聚体组合最终形成三叶草形状的肌酐水解酶,其六聚体及亚基结构如图1 [6]。肌酐水解酶结构为亚基A和B、C和D、E和F各自形成一个二聚体,每个二聚体内部亚基间(即亚基AB间、CD间或EF间)的接触面积约为1928 Å2,二聚体间亚基(即亚基BC间、DE间或FA间)的接触面积约为1473 Å2。
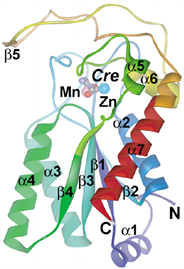
Figure 1. Creatinine hydrolase hexamer structure and subunit structure
图1. 肌酐水解酶六聚体结构及亚基结构
肌酐水解酶的活性位点位于圆盘形六聚体酶的边缘。酶活位点由疏水性裂缝排列形成入口十分狭窄,这个疏水性裂缝由Trp174,Trp154,Tyr121,Phe182,Tyr153和Gly119组成,它们形成了底物结合位点。裂缝的底部存在着具有双核金属中心的催化位点。此外,双金属中心中的锌离子也与水分子接触共同催化底物发生反应 [6]。
Yamashita [7] 等人假设的肌酐水解酶催化肌酐水解为肌酸是一个具有两步过程的反应,首先,在第一步中,桥接在双核金属中心的水分子内部的氢氧根离子攻击肌酐的羰基碳;随后第二步中,四面体中间体稳定化之后,通过另一水分子同时使酰胺氮质子化来辅助碳–氮键的裂解。新形成的肌酸的羧酸盐基团与双核中心的两个金属离子结合。在产物释放后,活化的水分子恢复到初始状态,完成催化过程。
4. 肌酐水解酶的应用
目前肌酐水解酶主要应用于使用比色法的肌酐检测试剂盒中。早期的肌酐检测试剂盒主要通过Jaffé法进行检测,其特异性较差,易受到血清、尿液中非肌酐物质的影响导致测定的肌酐浓度偏高。随着酶法测定肌酐的方法被开发出来,Jaffé法已经被特异性更强的酶法所取代。在1979年 [8],就有酶法测定肌酐试剂盒被发明,其包含四种酶:肌酐水解酶、肌酸激酶、丙酮酸激酶和乳酸脱氢酶。血清或尿液中的肌酐首先被肌酐水解酶水解为肌酸;随后肌酸与ATP在肌酸激酶的作用下生成磷酸肌酸和ADP;ADP与PEP在丙酮酸激酶的作用下变为丙酮酸和ATP;最后丙酮酸与NADH+和H+在乳酸脱氢酶的作用下变为乳酸和NAD+。每1mol的肌酐按照此反应过程变为1 mol NAD+,在340 nm的紫外波长下测定生成的NAD+的吸光度值即可测定肌酐浓度。
随后更多新型的肌酐检测试剂盒被发明出来,目前使用较多的为含有肌酐水解酶、肌酸水解酶、肌氨酸氧化酶和过氧化物酶的试剂盒。肌酐在肌酐水解酶的作用下水解为肌酸;随后肌酸被肌酸水解酶水解为肌氨酸;肌氨酸又被肌氨酸氧化酶催化为甘氨酸、过氧化氢;最后过氧化氢与4-氨基安替比林和2,4-DCP在过氧化物酶的作用下生成紫红色化合物醌亚胺,在546 nm处测定其吸光度值后,测定标准和空白样品后即可算出肌酐含量 [9]。
目前也有将肌酐水解酶固定在铂电极上制成电极以检测肌酐的方法,Yadav等 [10] 通过将肌酐水解酶、肌酸水解酶和肌氨酸氧化酶一同固定在氧化铁纳米颗粒/壳聚糖接枝聚苯胺复合膜,通过戊二醛偶合电沉积在铂电极表面。与上述酶法测定肌酐方法类似,每个肌酐在肌酐水解酶、肌酸水解酶和肌氨酸氧化酶的作用下产生过氧化氢,并在电压作用下分解产生两个电子,电子进入铂电极。测定时使用恒电位仪/恒电流仪连接工作电极,以Ag/AgCl作为参比电极,以Pt线作为辅助电极,构成肌酐生物传感器,通过固定电压,测定电流大小计算肌酐浓度。对比Jaffé法及酶法,使用电极可以改善生物传感系统的灵敏度,稳定性和抗干扰能力,从而使肌酐测定更加准确、可靠,并能够降低检测成本。
5. 总结与展望
肌酐水解酶出现于微生物,对微生物获取氮源具有一定意义。目前为止,肌酐水解酶主要应用于血清、尿液中肌酐的测定,但酶法测定肌酐仍有不足之处。而最新研究的酶电极具有较强的灵敏度,稳定性和抗干扰能力,具有广大的使用前景。
NOTES
*通讯作者。