1. 前言
肝癌作为一种恶性程度高而生存预后差的恶性肿瘤,其发病机制复杂,原因多样化 [1],且发病率在全球范围内多年保持居高不下的态势 [2]。2020年的global cancer statistics中的数据显示,肝癌发病率位居全球第六 [3];DFNA5基因全名为耳聋相关肿瘤抑制因子(Deafness Tumor Inhibitor Suppressor, DFNA5) [4],又名GSDME (Gasdermin E),Gene ID:1687,是GSDM家族成员,早期研究集中于家族遗传性耳聋 [5] [6],后期研究发现,DFNA5基因可能与肿瘤炎性环境的形成有关 [7] [8] [9],其可通过自身蛋白剪切活化,引发肿瘤细胞打孔功能而展示出肿瘤抑制潜能 [9] [10]。也有研究集中探讨DFNA5甲基化与肿瘤的关系 [11] [12] 以及DFNA5作为GSDME蛋白可能参与不同细胞死亡方式的相关实验研究 [13] [14],包括细胞凋亡 [14],铁死亡 [15] 或继发性坏死 [16] 与细胞焦亡的死亡形式之间的转化,甚至于GSDME基因直接参与细胞焦亡的发生过程 [17] [18] [19],大量实验研究论证了DFNA5基因的甲基化与结直肠癌 [20],胃癌 [21] 及乳腺癌 [22] [23] 之间的关系,少有研究探讨其在肝癌中的表达及其与预后之间的关系。
生物信息学是随着计算机科学中的软硬件升级高速发展的学科,适当利用生物信息学有利于加速基础实践研究的早期进程,而现有的研究内容中鲜有见到对DFNA5基因与肝癌关系预测的相关研究。本研究使用生物信息学,利用BioGPS、HPA、Oncomine、TCGA、CCLE等数据库,对DFNA5基因在人体组织及肿瘤组织中的表达分布进行分析,进一步对其在肝癌及实验肝癌细胞系中的相关表达进行综合分析,最后分析DFNA5基因表达量与肝癌患者的生存预后之间的关系,为后期实验研究寻找敏感性更高,特异性更强的早期诊断指标以及治疗靶点。
2. 资料与方法
2.1. 在线网络数据库数据资料收集与整理
1) 在BioGPS数据库中搜索“DFNA5”,选择Gene ID为1687的人源GSDME作为筛选对象。
2) 在Human Protein Atlas (HPA)数据库中检索“DFNA5”基因,提取其作为Gasdermin E在肝癌中的表达情况和细胞中的蛋白质定位情况。
3) 在Oncomine数据库中搜索DFNA5基因(Gene ID:1687),筛选Analysis Type:Cancer vs Normal Analysis;Cancer Type:Liver Cancer;Data Type:mRNA,收集DFNA5基因在不同肝癌研究中的表达并对结果进行集中分析。
4) 在TCGA数据库中检索下载肝癌的转录组数据,统计分析DFNA5基因在癌组织和癌旁组织中的表达差异。
5) 在CCLE中搜索DFNA5基因,下载其在各类肿瘤细胞中的mRNA表达数据。提取DFNA5在肝癌细胞中的表达数据并进行统计分析;下载并收集DFNA在各类肿瘤细胞中的DNA甲基化数据进行进一步统计分析。
6) 在Kaplan Meier-Plotter数据库中选择“Liver cancer”条目,Gene symbol:DFNA5;筛选“Gender”,分别收集“Male”,“Female”相关结果。
2.2. 统计分析
所有数据采用GraphPad Prism软件进行统计分析。DFNA5基因的表达与肝癌的预后的关系采用Kaplan-Meier模型分析,并且绘制生存曲线。以P值 < 0.05作为统计学标准,存在统计学意义。
3. 结果
3.1. DFNA5在人体中的整体表达情况
3.1.1. BioGPS数据库
在BioGPS数据库中搜索DFNA5基因,进行在线分析,结果如图1所示,DFNA5基因在人体绝大部分组织中表达,而其中,在肝脏组织中的表达水平为14.65+/−0.106略高于整体表达水平的中位值:Median = 12.1。
3.1.2. HPA数据库
在HPA数据库中搜索DFNA5基因,其中对TCGA数据在线分析结果提示,DFNA5基因组织特异性低;GSDME作为耳聋相关肿瘤抑制因子,可能是肝癌的不利预后指标,并且其可能通过与TP53联合作用,影响到TP53参与调节的针对DNA损伤的细胞应答 [24]。对数据库中的多种肿瘤组织中的DFNA5的表达情况进行综合分析,RNA表达状况统计包含17种常见肿瘤组织的数据,如图2所示,DFNA5基
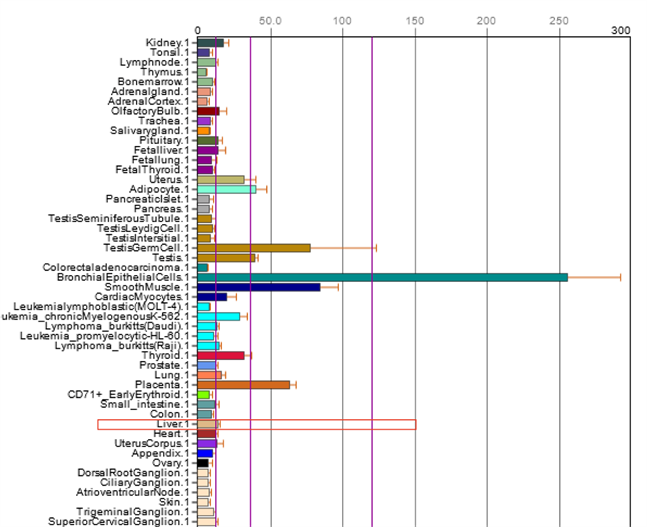
Figure 1. Differential expression distribution of DFNA5 in human tissues
图1. DFNA5在人体组织中的差异表达分布
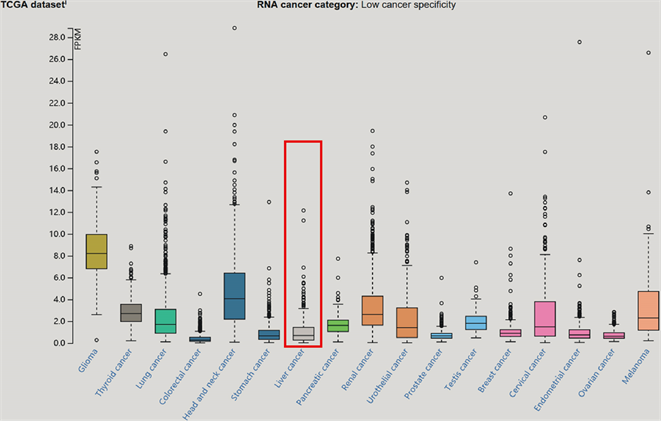
Figure 2. RNA expression in various tumor tissues DFNA5
图2. 不同肿瘤组织中DFNA5 的RNA的表达情况
因在其中均有表达,DFNA5的RNA表达水平在17种常见癌症中处于第13位;对DFNA5在蛋白水平的表达进行统计,如图3所示,GSDME蛋白在肝癌中的表达强度位于第六位;在免疫荧光图片中选择“Antibody”、“Nucleus”和“ER”,联合模型示意图4(a),图4(b)可见,DFNA5的蛋白表达主要分布于细胞质基质中。
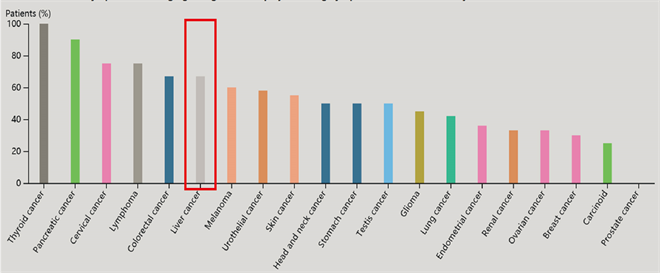
Figure 3. The expression of DFNA5 protein in various tumor tissues
图3. DFNA5蛋白在各类肿瘤组织中的表达情况
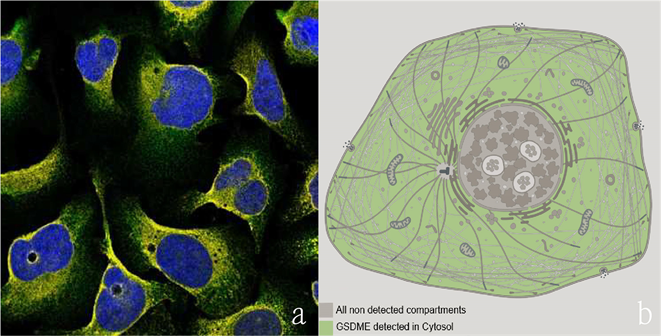
Figure 4. (a) Immunofluorescence staining of DFNA5 protein in human cells (for example, U-2 OS cells, the antibody is HPA1326 showing green fluorescence, rabbit anti-DFNA5; nucleus stained blue, endoplasmic reticulum stained yellow); (b) DFNA5 protein localization in the cell Schematic model diagram
图4. (a) 人类细胞中DFNA5蛋白免疫荧光染色(例:U-2 OS细胞,抗体为HPA1326显示为绿色荧光,兔抗DFNA5;细胞核染蓝色,内质网染黄色);(b) DFNA5蛋白在细胞中定位示意模型图
3.2. DFNA5在肝癌中的分布
3.2.1. Oncomine数据库
在Oncomine数据库中进行分析发现,DFNA5 mRNA在肝癌组织和正常肝组织之间的表达差异的相关研究为包含了678个数据样本的4项研究。针对这4项研究在Oncomine数据库中进行综合分析发现,DFNA5 mRNA在肝癌组织中的表达量高于在正常肝组织中的表现(见图5),该差异具有统计学意义(P = 0.004, Median Rank = 3109.0)。
3.2.2. TCGA数据库
TCGA数据库中比较肝癌与正常肝组织中DFNA5的mRNA数据表达差异,结果可见图6,DFNA5的mRNA表达相对于正常肝组织,在肝癌组织中表达量更高,P < 0.05,有统计学意义。

Figure 5. Comprehensive expression analysis of DFNA5 in liver cancer research (Oncomine database)
图5. DFNA5在肝癌研究中的综合表达分析(Oncomine数据库)
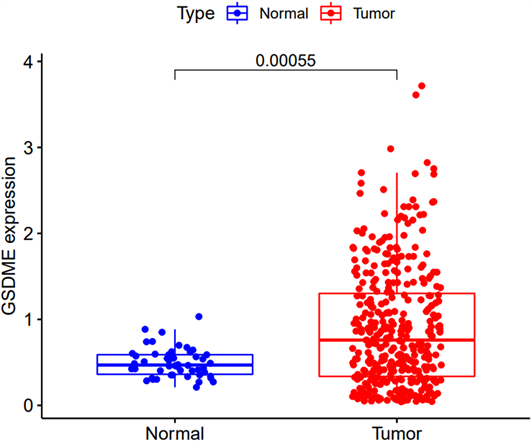
Figure 6. DFNA5 mRNA expression status in liver cancer (TCGA database)
图6. 肝癌中DFNA5的mRNA表达状况(TCGA数据库)
3.3. DFNA5 mRNA在CCLE的不同肿瘤细胞系中的表达分布
DFNA5 mRNA在CCLE的不同肿瘤细胞系中的表达分布,在CCLE数据库中搜索DFNA5,结果显示,DFNA5的mRNA在不同肿瘤细胞系中表达状态不一;肝癌细胞系中DFNA5 mRNA高表达,且表达量为肿瘤细胞系中排名第12位(见图7)。
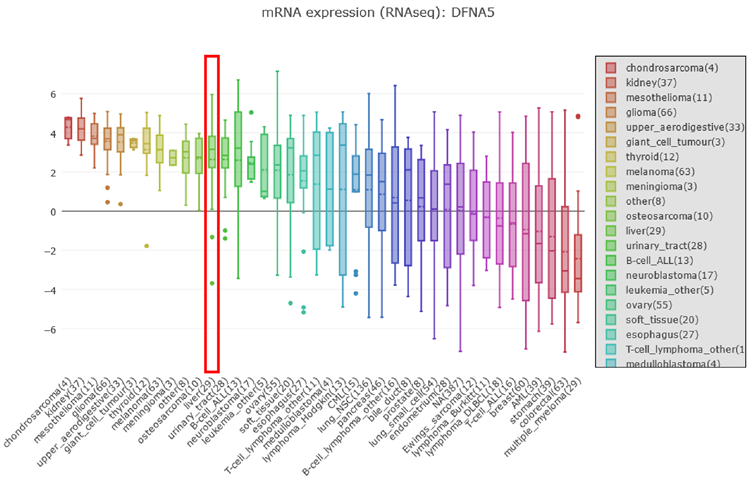
Figure 7. Distribution of DFNA5 mRNA in different tumor cell lines
图7. DFNA5 mRNA在不同肿瘤细胞系中的分布
3.4. DFNA5的DNA甲基化状态在CCLE的不同肿瘤细胞系中的汇总分析
CCLE数据库中搜索DFNA5,选择数据类型(DNA methylation RRBS: DFNA5),DFNA5的DNA甲基化程度分布在不同肿瘤细胞系中不同;以结直肠癌为例,其DFNA5的mRNA表达量处倒数第二,但DNA甲基化程度处第二位,提示大多数细胞系中DFNA5的DNA甲基化与mRNA的表达可能是负相关;肝癌细胞系中DFNA5的甲基化程度有限,且38种肿瘤细胞系中排名第24位(见图8)。
3.5. DFNA5对肝癌患者预后的影响及其性别分析
在Kaplan-Meier-Plotter数据库中 [25],选择Liver,对其中包含的364例肝癌患者的生存数据进行分析,如图9,DFNA5的表达水平对肝癌患者的生存预后有显著影响。DFNA5的高表达是肝癌患者生存预后的不利指标,(P < 0.05):且DFNA5的表达对生存预后的影响具有明显的性别差异, DFNA5基因的高表达对于女性的生存预后影响显著,在女性患者中高表达组的生存率显著降低,P = 0.00013,而在男性患者中特别是在长期生存后进行数据统计发现DFNA5的高表达对于男性肝癌患者生存率影响有限,P = 0.051,从统计数据上来看,DFNA5基因的表达量在女性肝癌患者中对生存预后的影响更为显著。
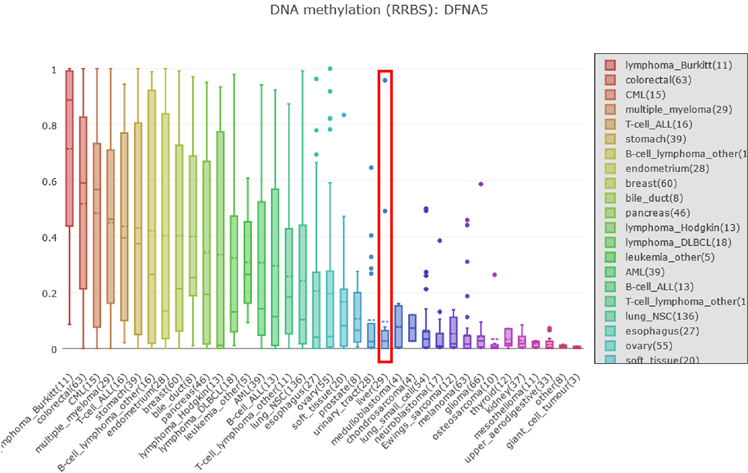
Figure 8. Degree of methylation DFNA5 DNA in different tumor cell lines distribution
图8. DFNA5在不同肿瘤细胞系中的DNA的甲基化程度的分布
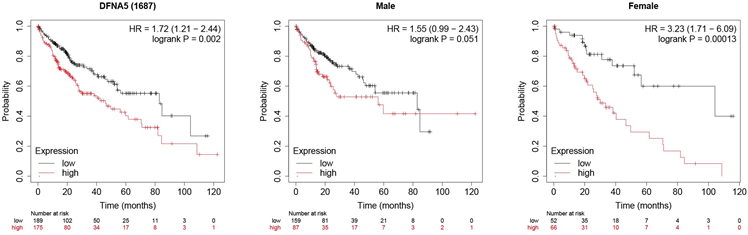
Figure 9. DFNA5 expression level and overall survival rate of the patients survival curve
图9. DFNA5表达量与患者总体生存率生存曲线
4. 讨论
肝癌作为我国常见的恶性肿瘤,其死亡率位居全球肿瘤死亡率的第三位,且肝癌是一种恶性程度非常高的疾病,发病机制由多种基因共同调控,肝癌形成的原因十分复杂且可能包括多种因素在不同阶段的共同作用。
DFNA5基因是家族遗传性耳聋的关键基因,近些年研究发现DFNA5蛋白的N端结构域是细胞焦亡发生的一种关键蛋白,DFNA5的N端蛋白可导致细胞膜性结构打孔,诱导细胞焦亡的发生,从而参与机体的某些生理或病理进程。而相关研究发现,细胞焦亡进程在肿瘤细胞中受到抑制,对于肿瘤的形成与发展有重要意义 [10]。目前对于焦亡的研究迅速开展,但大多基础实验专注于DFNA5基因在肿瘤细胞参与细胞焦亡的发生或介导细胞从不同死亡形式向细胞焦亡的转化过程 [13] - [18],有研究指出GSDME (DFNA5)可通过启动焦亡进程后激活抗肿瘤免疫抑制肿瘤的生长,对于肿瘤周围炎性环境的形成有重要意义 [7] [8] [9] [10] [20];而DFNA5与肿瘤关联的相关研究集中于DFNA5甲基化 [11] [12] 与乳腺癌,结直肠癌,胃癌的发生发展转移的关系 [21] [22] [23] [24],有研究提出DFNA5基因参与P53协同作用的肿瘤细胞抑制作用 [19]。
本研究通过对公共数据库中的焦亡相关基因DFNA5的相关数据进行挖掘统计荟萃分析,首先在BioGPS中分析发现DFNA5基因在正常组织中的分布表现无显著组织特异性,而通过在HPA数据库中进一步分析发现DFNA5在细胞中的相关定位表现为编码的蛋白广泛存在于细胞质基质中,且DFNA5在不同肿瘤中表达情况不一,其蛋白表达在甲状腺肿瘤中表现比例最高,肝癌表达则位于第6位;核酸表达水平上,则是在神经胶质瘤中最高,在肝癌中的RNA表达水平与大多数肿瘤类型无显著区别。综合多项数据库分析发现,DFNA5基因在肝癌与正常肝组织中的表达存在一定差异,DFNA5在大多数肝癌中表现为高表达,可以对其在肝癌发生发展机制中的作用做进一步研究。基于Kaplan Meier在线网络对364名肝癌患者进行生存预后分析,发现DFNA5基因高表达时,患者的生存时间缩短,且与患者的性别分布有关,DFNA5的表达水平对于女性患者的生存预后有显著影响,DFNA5高表达的女性患者相对于低表达组,总体生存率明显降低,提示DFNA5直接表达对于肝癌预后的影响,可能与性别之间存在某种联系,在进一步研究中可以考察该基因的表达与性别相关部分表达或指标之间的联系。总而言之,DFNA5在一定程度上可能影响肝癌的发生发展与治疗预后,在不同肿瘤组织中表达状况不同,在肝癌中高表达,但表达相对于其他肿瘤没有组织特异性,但是DFNA5高表达直接影响女性患者的总体生存预后。因此,DFNA5不适宜作为肝癌诊断的特异性肿瘤标志物,但存在作为女性肝癌患者治疗后生存预后的检测指标的潜力。
DFNA5对于肿瘤发生发展变化的影响机制有待进一步探索发掘,其在不同肿瘤中的表达影响有区别。深入研究DFNA5基因相关分子机制与信号通路,通过不断完善通路实验研究,期望在后续研究支持下找到高敏感度,特异性强的肿瘤早期诊断指标与治疗靶点。
基金项目
国家自然科学基金项目(编号:81872491);重大新药创制国家科技重大专项(2018ZX09733001-004-002);广西科技研究基地和人才专项计划(AD18126021);区域性高发肿瘤早期防治研究教育部重点实验室(GKE2018-03, GKE2019-09)。