1. 引言
弹性意味着能够适应外部条件的改变,迅速从崩溃中恢复 [1]。Ayyub进一步将系统的弹性定义为“面对干扰等不确定情况时系统保持其功能和性能的持久性” [2]。网络弹性在许多领域都有重要应用。具体到不同网络,系统的弹性也有不同含义。比如,我们常见的社会网络中,弹性是指抵御社会行为变化的能力,它度量了特定人群和动物的社会行为的稳定性;工程网络的弹性(传统意义下的“工程弹性”)衡量的是系统受干扰之后恢复到稳定状态或平衡点的时间;生物网络中的弹性是指机体在面临外界环境参数变化时保持正常运作的能力,它能够起到阻断或延缓疾病进程的作用。
根据动力系统分岔理论,疾病的发展过程可以分为3个阶段/状态:a) 正常状态,它是系统远离分叉点的稳定状态,其势能低,弹性高,此时系统抵御外界干扰的能力较强;b) 前疾病状态,可视作相变前的临界点,其势能高,弹性低,此时外界微小的变化就可能使系统迅速进入疾病状态;c) 疾病状态,是系统经过分岔后的另一种稳定状态,此时系统又会回复到势能低,弹性高的情况 [3] [4]。其中,正常状态和前疾病状态均为可逆过程,而疾病状态是不可逆的(图1A)。因此,识别前疾病状态对于防止疾病恶性突变,了解疾病发生与演化至关重要。注意到弹性在正常状态和疾病状态较高,在前疾病状态较低的特点,我们可以利用生物网络弹性来识别前疾病状态。
基因关联网络是一种常见的生物网络 [5],它是以基因为节点,以基因之间的相互关联为边构成的系统。最简单的基因网络生成方法是计算基因数据集的皮尔森相关系数矩阵,如果两基因的相关系数低于某阈值,则认为它们之间没有边连接;反之,这对基因之间有边连接。由此得到的网络被称作“相关性网络”。然而,这样的网络难以区分基因间的直接相互作用和间接相互作用 [6],因为基因间的高相关系数指示的相互作用可能是直接的,也可能是间接的。为了弥补这一缺陷,许多研究材料在PLS的基础上计算了基因间的连通性得分,并用连通性得分来衡量基因与基因的直接相互作用强度,进而构建基因关联网络(PLS网络) [6] [7] [8]。在本文中我们采用了Gill et al.的方法 [7] 构建基因关联网络。
从高通量的基因数据中重构出基因网络后,我们面临的一个极具挑战性的问题是如何量化网络弹性。在度量网络弹性方面,Wang et al. [9] 使用系统的恢复能力来度量网络弹性;Ayyub假定破坏性事件发生服从泊松分布,构建了网络弹性的数学模型 [1];Yalda以华盛顿地铁为案例构建交通网络 [2],在Ayyub提出模型的基础上,通过间接评估网络效率得到了弹性指标;Jianxi Gao et al. [10] 研究了复杂网络中弹性的普遍特征,结果表明网络拓扑指标中网络密度、非均质性和均匀性与网络弹性密切相关。然而,这些量化网络弹性的方法大多基于模型,并且仅适用于低维度的数据,对于高通量的基因数据并不适用。因此,找到一种可靠、高效的方法度量基因关联网络弹性的需求十分迫切。
在本文中,我们提出了一个基于交叉熵的计算网络弹性的方法。具体来说,首先要根据基因表达数据和基因间的皮尔森相关系数构造每个基因的分布,接着计算两两基因分布的交叉熵,最后结合先前构造的PLS网络,计算网络弹性指标(图1B)。为了便于探测疾病恶性突变前的临界点,我们将得到的网络弹性指标取逆,得到指标U,这样U的峰值将对应着我们预测出的临界点(图1C)。为验证方法的有效性,
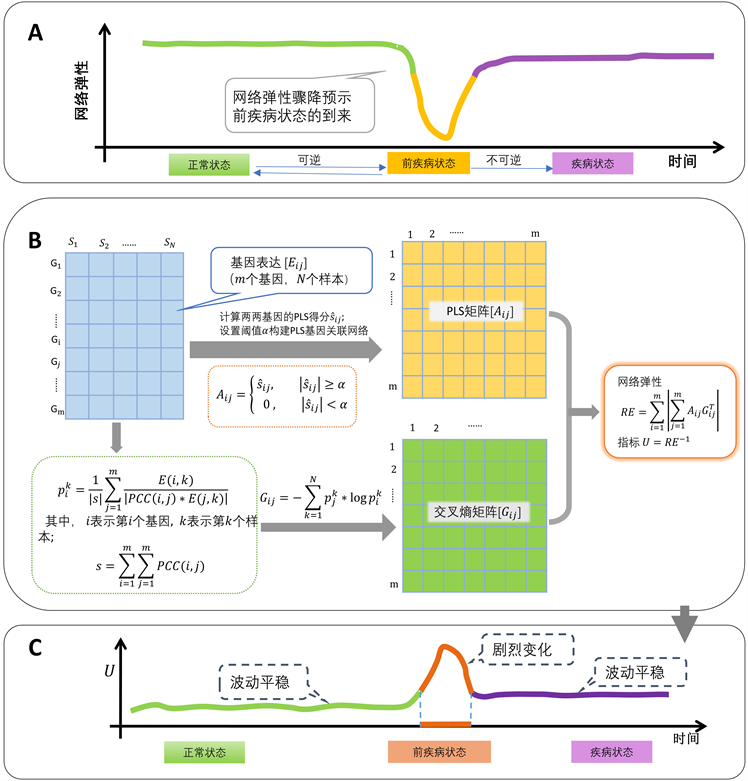
Figure 1. (A) The changing trend of network resilience during the progression of complex diseases (B) The algorithm of evaluating network resilience (see more details in Section 3) (C) Property of indicator U during the progression of complex diseases
图1. (A) 复杂疾病发展过程中的网络弹性变化趋势图;(B) 计算网络弹性的算法流程(详见第三节);(C) 复杂疾病发展过程中指标U的特性
我们将其应用于1个模拟数据集和5个真实数据集中,结果显示,前疾病状态可以被快速、准确地识别出来。总的来说,基于网络弹性的临界点预测方法的计算复杂度小,抗噪性能好,并且,由于它是一种无模型的方法,所以不需要对训练数据进行任何学习,这使得它对高通量基因组数据的适用范围更广。另外,我们提供了一种全新的网络弹性的度量方式,它可以用来描述疾病发展过程中的网络结构差异,为从网络视角理解疾病的发生与演化提供新的思路。
本文的组织结构安排如下:第二节是基于网络弹性的临界点预测在仿真数据集和4个真实数据集中的应用;第三节是总结与展望;第四节将详细介绍计算网络弹性的生物学方法。
2. 实验结果
2.1. 在模拟数据集上的应用
我们先用16节点的模拟网络来验证方法的有效性(图2A)。该网络是一组16个基因的调控表示,其中16个微分方程代表16个基因的调控机制。此模型用Michaelis-Menten形式表示。这类调控网络通常被用于研究遗传调控,包括转录和翻译过程 [11] [12] [13],以及多稳定性和非线性生物过程 [14] [15]。此外,Michaelis-Menten形式的分叉经常被用来模拟基因调控网络的时期转移 [16] [17]。此模型的参数q在−0.5到0.15之间变化。
我们首先使用上述模拟数据集,通过Cytoscape软件(http://www.cytoscape.org/)构建PLS网络,图2B给出了部分时间点对应的PLS网络。从图中可以清楚地看到,正常状态的网络中的边都比较密集,网络结构更稳定;而前疾病状态的网络表现为两个局部网络相接,网络中的边也明显更稀疏,这样的结构稳定性更差。也就是说,前疾病状态与正常状态和疾病状态的PLS网络拓扑结构差异显著,因此PLS网络邻接矩阵可以成为我们衡量网络弹性的一个有力工具。
在得到每个时间点的PLS矩阵后,我们再结合对应时间点的交叉熵矩阵,计算出各时间点的指标U。U随参数q的变化趋势如图2B所示。从图中可以看到U的峰值出现在q = 0时,这表明q = 0预示着相变点的来临,也表明我们预测的临界点与实际观测一致。
2.2. 在真实数据集上的应用
我们将基于网络弹性的临界点预测方法应用于5个真实的数据集。其中包括源于NCBO的GEO数据库的小鼠肺部急性损伤基因表达(GSE2625)数据集(https://www.ncbi.nlm.nih.gov/),以及来自TCGA数据库的肺腺癌数据集(简称LUAD)、胃腺癌数据集(简称STAD)、甲状腺癌数据集(简称THCA)、结肠癌数据集(简称COAD) (http://cancergenome.nih.gov)。
选取富含信息的基因是降低数据复杂度、提高信噪比的第一步 [18]。因此在应用网络弹性预测疾病发展过程中的临界点之前,我们要对数据集进行预处理,其目的是筛选出在不同实验条件下变化显著的基因。预处理的步骤如下:
a) 2-fold-chage:计算各基因在实验组样本中的标准差和在对照组样本中的标准差,如果前者至少是后者的2倍,则将该基因记录下来;
b) t-test:对每个时间点实验组和对照组的样本进行显著性水平为0.05的t检验,筛选差异程度最高的前300~400个基因;
c) 对每个时间点筛选出的基因取并集,形成新的数据集。
2.2.1. 识别小鼠肺部急性损伤的临界点
小鼠肺部急性损伤基因表达数据集(GSE2565)中,每个时间点可以探测到的基因个数为12,871个,
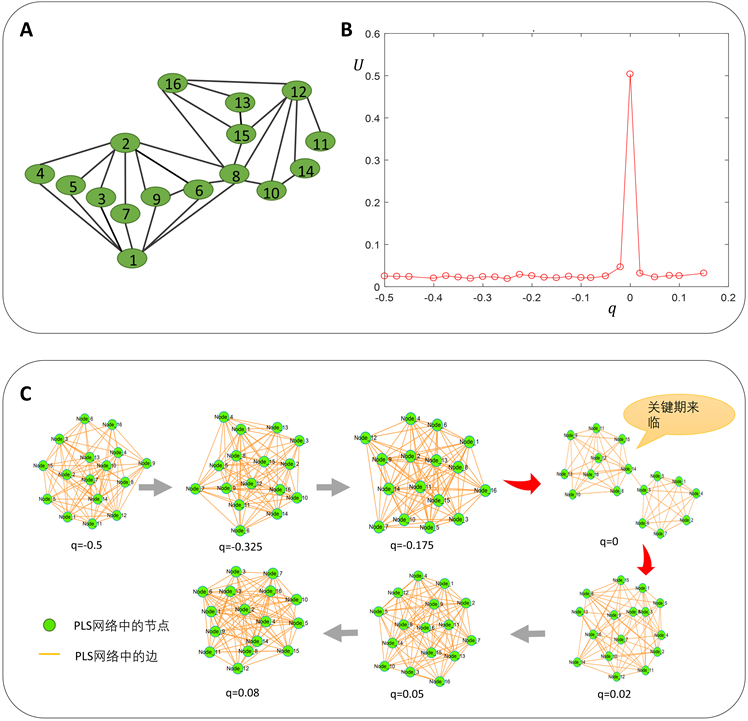
Figure 2. (A) 16-nodes gene regulation network based on numerical simulation (B) Curve: the indicator U changing with parameter q (C) 16-nodes PLS network at certain time points
图2. (A)基于数值模拟的16节点基因调控网络图(B)指标U随参数q的变化趋势图(C)部分时间点的16节点PLS网络
共 9个不同的采样点:分别是持续暴露于光气环境后的0小时,0.5小时,1小时,4小时,8小时,12小时,24小时,48小时,72小时。每个时间点设置的实验组和对照组中各有6个样本。实验结果表明,实验组的小鼠在吸入光气8小时后开始出现不同程度的肺部损伤,一直持续到第12小时。在第12小时,实验组的小鼠死亡率约为50%~60%,对照组的小鼠死亡率约为60%~70% [19]。
2.2.2. 识别癌症的临界点
该方法又被用于4个来自TCGA癌基因图谱的癌症数据集(肺腺癌、胃腺癌、甲状腺癌、结肠癌)。根据TCGA的临床资料,以上数据集由癌症样本和癌症近邻样本组成,同时各数据集中的癌症样本大部分被标记了不同的分期,这里,胃腺癌和肺腺癌总共有七个时期,甲状腺癌、结肠癌总共有四个时期。在上述4个数据集中,我们将未被标记分期信息的样本删去,把癌症近邻样本都看作参考样本。为了验证所识别的临界时期,我们对临界转化前和转换临界转化后的样本进行了Kaplan-Meier (Log-Rank)生存分析进行比较(图3C、图3E、图3G、图3J)。需要注意的是,临界转化前的样本的预后寿命通常比临界转化后的样本高。
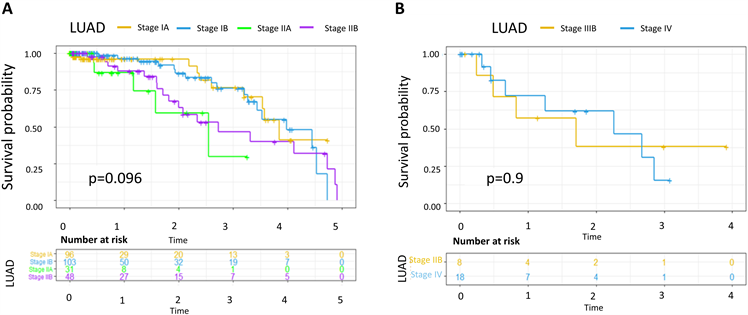
我们识别出的LUAD的临界期在IIIA期(图3B),也就是说,在此之后病人的癌症状况可能会迅速恶化。对LUAD IIIA期前后的样本分组进行Log-Rank检验,会发现IIIA期以前的样本的生存曲线和IIIA期之后的生存曲线存在显著差异(p < 0.0001,图3C)。并且,临界期之前(IA, IB, IIA, IIB)的生存曲线没有显著差异(p = 0.096,见附录),临界期之后的生存曲线差异显著性也不高(p = 0.9,见附录)。这些结果显示,LUAD病人在IIIA期后面临预后寿命的锐减,也表明IIIA期是癌症的一个关键期。同样地,我们可以判断STAD的临界期——IIIA期是一个关键期。
对于甲状腺癌(THCA),使用我们的方法识别出的癌症迅速恶化前的临界期为II期(图3D)。同时,我们发现,甲状腺癌II期之前和II期之后的生存曲线是显著不同的(p = 0.0045,图3E)。相应地,临界期之前样本的预计存活时间和要比临界期之后样本的预计存活时间长得多。另外,结肠癌(COAD)的临界期被确定在II期(图3F),并且COAD临界期之前和之后的生存曲线存在显著差异(p = 0.00059,图3G)。相应地,COAD中临界期之后的样本生存时间比临界期之后少得多。
上述结果表明,基于网络弹性的临界点预测方法可以识别出癌症恶化前的临界期,并且这些关键期和预后分析紧密相关。
3. 主要方法
3.1. 预备知识
3.1.1. 交叉熵
交叉熵的概念源于信息论,它可以衡量观测样本相对于参照样本的混乱程度。假定有两个概率分布:参照样本的分布为Q,
;观测样本的分布为P,
;那么观测样本与参照样本的交叉熵定义为 [20]:
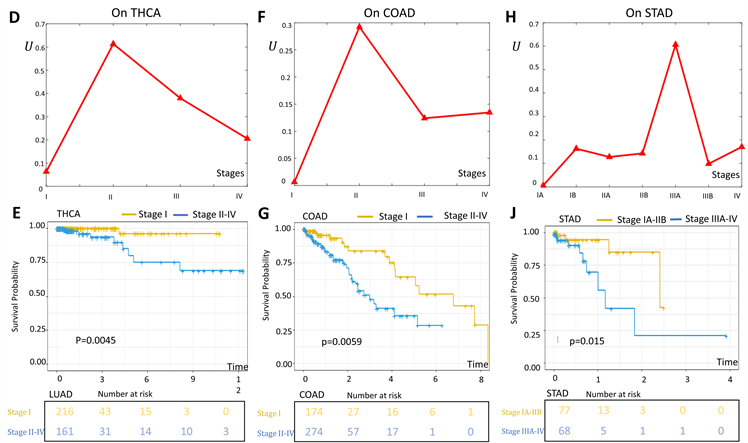
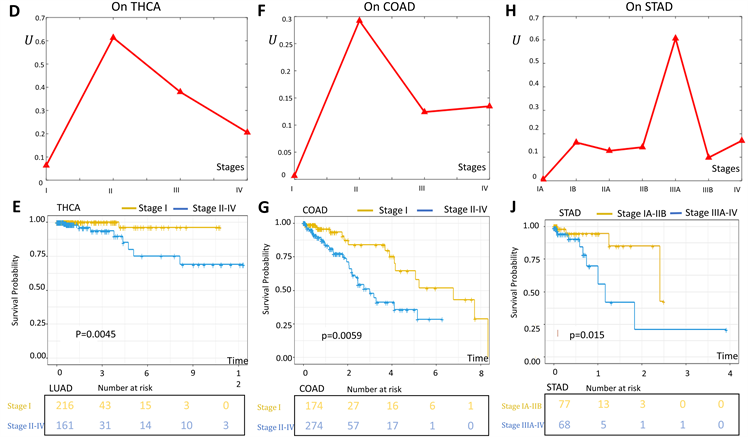
Figure 3. (A) Identifying critical stage on acute lung injury in mice (B) Identifying critical stage on LUAD (C) the survival curve before and after critical stage on LUAD (D) Identifying critical stage on THCA (E) the survival curve before and after critical stage on THCA (F) Identifying critical stage on COAD (G) the survival curve before and after critical stage on COAD (H) Identifying critical stage on STAD (J) the survival curve before and after critical stage on STAD
图3. (A)识别小鼠肺部急性损伤的临界点 (B)识别肺腺癌(LUAD)的临界点 (C)肺腺癌临界期前后的生存曲线 (D)识别甲状腺癌(THCA)的临界点 (E)甲状腺癌临界期前后的生存曲线 (F)识别结肠癌(COAD)的临界点 (G)结肠癌临界期前后的生存曲线 (H) 识别胃腺癌(STAD)的临界点 (J) 胃腺癌临界期前后的生存曲线
(1)
3.1.2.PLS (Partial Least Squares Regression)
PLS的核心思想是用每个基因的表达向量做偏最小二乘的拟合((2)式)。
表示标准化后的基因i的表达,
代表PLS的项数,
是
的线性组合 [7]。
(2)
利用PLS计算基因间相互作用强度的算法步骤如下:
步骤1:令
,
步骤2:计算
其中,
步骤3
,迭代计算
,如果
,则返回步骤2
最后,基因i和基因k的相互作用强度可以用下面的PLS得分来估计:
记
为PLS网络中基因i和基因k所连边的权,其中,
3.2. 理论基础
Baruch Barzel et al.研究了网络动力学中的普遍特征 [21],发现生物网络中节点 的扰动对其邻域内节点的影响可以表示为:
(3)
其中
表示网络的邻接矩阵,
表示基因j的扰动对基因i的影响程度。
表示节点i对其一阶邻居节点的影响力。
这里我们用基因j相对于基因i的混乱程度,即
来代替
((7)式)。
较大,意味着节点i微小的变动会对其邻域内节点产生很大影响力;那么反过来,节点i对它邻域内其他节点的变动不敏感。因此
可以看作是节点i维持其原有状态的能力。同时考虑到网络弹性由网络中各个节点刻画,我们将基因关联网络的弹性量化为:
(4)
3.3. 算法步骤
3.3.1. PLS网络构建
首先对基因表达数据Z-Score标准化,设置默认的PLS项数
,然后根据3.1.2节的算法计算PLS得分得到基因间边的权。
考虑到基因之间的关联网络结构通常是稀疏的,也就是说,大部分的潜在联系实际上是不存在的 [6],因此我们构建网络的方法是仅保留PLS得分较高的边(
)。具体做法是设置阈值参数
,参数
的确定方式是使得初始时间点的PLS网络中边数约为原始网络结构中的10% ((5)式)。
(5)
其中m为节点/基因个数,
是PLS矩阵,它按照下面的分段函数来确定:
(6)
3.3.2. 计算两两基因的交叉熵
:
的表达矩阵行为基因,列为样本
:
的交叉熵矩阵
:样本k中基因i的概率
(7)
(8)
(9)
(10)
3.3.3. 网络弹性测算
得到PLS矩阵和交叉熵矩阵后,按照(3)式和(4)式计算PLS网络的弹性。为了便于探测疾病恶性突变前的临界点,我们将得到的网络弹性指标取逆,得到不稳定性指标U,这样U的峰值将对应着我们预测出的临界点(图1C)
(11)
4. 总结与展望
网络弹性是复杂网络的关键特征之一,如何量化网络弹性一直是一个备受关注的问题。许多研究者已经在这方面做过大量工作,然而,他们方法大多基于模型,并且仅适用于低维度的数据,对于高通量的基因数据并不适用。在本文中,我们提出了一种数据驱动的衡量基因关联网络弹性的方法,并根据网络弹性在疾病发展过程中的特性来识别前疾病状态。为验证算法的有效性,我们将算法应用于1个模拟数据集和5个真实数据集中。结果表明,基于网络弹性的临界点预测方法可以快速、有效地识别复杂疾病恶性突变前的临界状态。
相对于传统的比较基因表达差异区分正常状态与疾病状态的方法,我们的方法旨在于捕捉疾病发展过程中的动态信息,识别前疾病状态。该方法有下面几个优点。首先,它是一种无模型的方法,只依赖于数据驱动,所以它对于高通量基因组数据的适用范围更广。其次,由于我们在构建基因关联网络时只保留PLS得分较高的边,所以使用该方法计算简单高效,而且可以减弱其受噪声的影响。最后,我们的方法可以用来描述疾病发展过程中的网络结构差异,为从网络视角理解疾病的发生与演化提供了新的思路。
基金项目
本文受广东省基础与应用基础研究基金资助(No. 2019B151502062)资助。
附录:
1. 数值模拟方法
首先考虑用下式表示的离散的时变动力系统:
(S1)
这里
是时刻k表示系统特征的n维的向量,
是控制系统参数缓慢变化的参数向量。
,该映射通常是非线性的。如果下面三个条件同时成立,那么带有参数 的动力系统发展方程(S1)存在分叉点或关键期[3]:
1、
是系统的不动点,即满足
;
2、存在
,使得一个或一对雅可比矩阵
的特征值等于1;
3、当
时,f的线性化系统的特征值不恒等于1。
以上3个条件说明系统在
经历了相变,或者说当P接近阈值
时系统出现分叉。当方程(S1)接近
,P值到达
前,我们假定系统处在稳定点,从而线性化系统的特征值范围都大于0小于1。P取值为
时,系统相变产生,这也意味着
为系统的分叉参数。
Michaelis-Menten形式调控网络通常被用于研究遗传调控,包括转录和翻译过程[11] [12] [13],以及多稳定性和非线性生物过程[14] [15]。此外,Michaelis-Menten形式的分叉经常被用来模拟基因调控网络的时期转移[16] [17]。本文中用到的模拟网络时用Michaelis-Menten形式表示的,它是一组16个基因的调控表示,网络中的节点表示生物分子,边表示生物分子之间正或负的调控关系。此模型的参数q在-0.5到0.15之间变化。下面16个微分方程代表16个基因的调控机制:
这里,q是控制参数,
是0均值的高斯噪声,
是mRNA-i的浓度,mRNA的降解速率是:
。系统的稳定平衡点是
。使用Euler函数,可以把微分方程组转化为
的形式:


其中,
是时间间隔,
是
在时刻k的向量。微分方程组的雅可比矩阵
,用
表示如下:
取
,从上式中解得16个不同得特征值:
显然,当q趋于0时,
趋于1。也就是说,
是系统开始变得不稳定的相变点,它是一个关键值点。控制参数q的取值范围是
。在每次数值模拟时,我们改变参数q的大小,把对应生成的10个样本作为参照样本,然后再根据每次模拟出的样本表达计算U的值。我们共进行了100次的数值模拟实验,将每次实验中得到的指标U取平均值,作为最终得分。最后,绘制出U和q的变化趋势图。U的峰值对应的q取值为0,与系统的相变点
重合,说明我们预测的临界点与实际观测一致。
2.LUAD临界期前后的生存曲线
我们对LUAD IIIA期前后的样本分别进行Log-Rank检验。p可以用来衡量生存曲线的差异程度,p值越接近0,表明差异程度越大。图A为肺腺癌(LUAD)临界期前的生存曲线,其中黄色,蓝色、绿色和紫色的曲线分别表示肺腺癌患者IA期,IB期、IIA期和IIB期的生存曲线。从图中可以看出,临界期之前(IA, IB, IIA, IIB)的生存曲线并没有显著差异(p = 0.096)。图B为肺腺癌(LUAD)临界期后的生存曲线,黄色曲线为肺腺癌患者IIIB期的生存曲线,蓝色曲线为肺腺癌患者IV期的生存曲线。生存分析结果表明,p = 0.9,即肺腺癌患者IIIB期和IV的生存曲线差异程度较小。