1. 引言
对医学图像处理的应用是现代医学分析中的重要部分,其能够有效地辅助医生进行病情判断,提高诊断效率,使用机器学习方法对医学图像进行高效地分析 [1] 有着广泛的实际应用价值。医学图像分析包括医学图像分类、医学图像分割等,其中医学图像分类是基础任务,旨在自动识别和分类不同类型的病变、组织或器官。近些年来,研究⼈员开发了大量医学图像分类方法 [2] [3] [4] 。这些方法可以减少⼈工分类所需的时间和精力。现在大多数方法均是基于深度学习的数据驱动的有监督学习方法,对标注数据的数量和质量有着极高的要求。然而,目前已有的医学图像分类的数据集规模都不大,这是因为医学图像的标签需要专业的医生来标注,成本较高。此外,在硬件条件有限制的情况下,设备生成的医学图像可能会导致噪声和模糊效果,使得图像质量降低,因此需要更有效的特征表示建模来实现稳健的医学图像分类。
不同于有监督学习,自监督学习不需要标签数据,而是通过构造自监督任务,利用海量无标签数据进行预训练,然后应用于各种下游任务上。在自然图像理解中,自监督学习的方法已经可以在多种不同的下游任务上达到领先的性能 [5] - [10] 。尤其是近些年来,基于对比学习的方法 [5] [6] [7] [8] 和基于图像掩码建模 [9] 的方法,都通过在大量无标注数据上进行预训练,在图像分类、目标检测、图像分割、图文检索等多个下游任务上追平甚至超越有监督方法。自监督预训练的方法优势在于不需要有标注数据,训练数据集的规模不受标签数据数量的限制,从而可以训练一个稳健的图像表征,在下游任务中取得更高的精度。因此,自监督表征学习非常适合于医学图像分类这种标注数据匮乏的任务。不同于自然图像,医学图像的分类对图像整体结构的理解和病灶区域处的细节纹理信息识别精度要求很高。然而,如图1所示,现有的方法为了设计高效的自监督任务,或是对图像进行高维的压缩表征 [5] [6] [7] [8] ,或是对图像进行大面积掩码 [9] ,上述两种信息的损失很大。
为了为医学图像分类提供一个更稳健的图像特征表示,关键在于要保证图像结构与全局信息、细节纹理信息的完整性,避免在进行预训练时图像信息损失过大。除了基于对比学习和基于图像掩码建模的两类主流方法之外,最近有研究人员也在探索将扩散模型用于自监督图像表征学习 [10] 。扩散模型训练多级去噪的过程可以看作是在训练一个多级的去噪自编码器 [11] 。通过识别出图像中的高斯噪声并重构出干净图像,去噪自编码器能够提取出更稳健地提取图像特征。因此,将去噪扩散模型用于图像表征学习是一种自然的想法。扩散模型是一种图到图的模型,并且没有对图像进行大面积的掩码,保持了完整的图像全局信息和细节纹理信息,相较于现有的基于对⽐学习和基于图像掩码建模两类自监督学习方法,更适合用于医学图像的表征预训练。

Figure 1. Diagram of the practical teaching system of automation major
图1. 三类自监督表征学习图示
本文提出了一种基于扩散模型自监督表征学习的医学图像分类方法MicDiffRep,用于为医学图像分类训练一个文件的图像特征提取器。MicDiffRep能够在预训练阶段尽可能地保证图像全局信息和细节纹理信息不丢失,学习到医学图像的整体结构和纹理细节,从而在下游的医学图像分类任务上取得更好的表现。扩散模型的具体结构一般是一个图到图的卷积神经网络(Convolutional Neural Network, CNN),在对图像进行卷积处理的过程中,模型会更多地关注于图像的局部信息。为了提高模型对图像全局信息的利用,本文提出了一个多尺度特征聚合模块MSFA,将来自不同层,不同尺度的特征进行聚合用于医学图像分类。实验结果显示,在脑瘤图像分类任务上,本文提出的MicDiffRep大幅领先其他现有自监督表征模型。
2. 方法
2.1. 扩散模型背景
扩散模型 [12] [13] [14] [15] 最初被提出用于图像生成任务。训练时,通过对干净图像添加不同程度高斯噪声,然后训练一个去噪模型来识别出带噪图片中的噪声,从而在推理生图时能够从一个随机采样的高斯噪声图中,一步步去噪,最终得到一张新的图像。扩散模型在不同的时间步t生成不同强度的高斯噪声,从而定义出一个高斯噪声序列
,其中αt,σt是手工设定的超参数,用于控制不同时间步加在干净图片x0上的高斯噪声强度。当T足够大时,带噪图像近似为一个完全噪声的高斯分布
。扩散模型的训练目标就是去噪,即预测加到干净图片上的高斯噪声。具体来说,是训练一个参数化网络εθ,在每个时间步t,根据当前步的带噪图像xt,预测前一步t − 1的高斯噪声均值
,高斯噪声的方差
是常数,不需要预测。也就是说,在每一个时间步t预测其上⼀步的带噪图像xt − 1:
(1)
上述扩散和反向过程可以等价于训练⼀个去噪自编码器:
(2)
正是因此,本⽂提出的MicDiffRep方法将扩散模型视作是⼀个多级的去噪自编码器,为医学图像分类提取完整、稳健的特征表示。
2.2. MicDiffRep
图2展示了本⽂提出的MicDiffRep方法的整体框架。输⼊在时间步t的带噪医学图像xt及时间步嵌⼊et,输出是当前时间步模型预测的噪声εt。扩散模型的参数化网络εθ是UNet网络 [12] [13] [14] 或视觉Transformer网络 [15] ,为了兼顾模型的性能和训练效率,MicDiffRep选用UNet网络作为基础的网络结构。
MicDiffRep方法的训练分为预训练和线性微调两个阶段。在预训练阶段,在大规模无标注的医学图像数据上训练扩散模型,损失函数即(2)式。在线性微调阶段,将UNet不同层,不同尺度的图像特征提取出来,用于医学图像分类。使用交叉熵分类损失来驱动模型的训练:
(3)
其中,N是样本的数量,yi,yi是分别是模型预测的第i个样本的标签和该样本的真实标签。

Figure 2. Diagram of the practical teaching system of automation major
图2. MicDiffRep方法整体框架
2.3. 多尺度特征聚合模块MSFA
受到CNN局部性归纳偏置的限制,UNet网络的浅层特征难以总览图像的全局信息,而深层特征则的细节纹理信息有所损失。为了保持图像的全局信息和细节纹理信息,我们在MicDiffRep中引⼊了一个多尺度特征聚合模块MSFA,来融合UNet网络的深层特征和浅层特征。MSFA模块的工作流程如图3所示。首先,将UNet中不同层的特征进行拼接:
(4)
然后经过一个映射层和激活函数ReLU对拼接特征进行深度聚合,得到聚合特征Fagg。在完成特征聚合之后,Fagg再通过映射头,和Softmax层之后输出各类别的分类概率:
(5)
(6)
其中,W,M分别是两个映射层的参数矩阵。
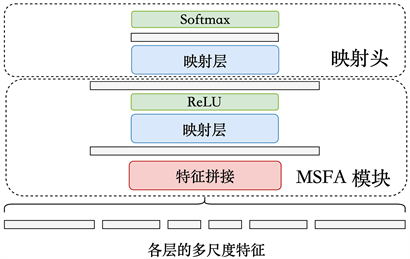
Figure 3. Diagram of the practical teaching system of automation major
图3. MSFA多尺度特征聚合模块
3. 实验结果与分析
3.1. 数据集
在预训练阶段,由于不需要任何标注,所以可以使用网络上海量的医学图像数据作为训练数据。在实验中,我们收集了共约80,000张医学图像用于预训练。
本文使用脑瘤图像数据集Crystal Clean数据集进行线性分类实验,该数据集共包含四个类别,分别是Normal,Glioma,Meningioma和Pituitary,共21,880张图像,其中训练集共17,533张图像,测试集4347张图像。
3.2. 实验环境及实验参数
我们在机器学习图形工作站上进行实验,配备CPU i5-12700F,内存DDR5 6000Hz 16 G,GPU为NVIDIA GeForce RTX 4070,显存12 GB。软件环境:操作系统Ubuntu 18.04 LTS,CUDA 12.1,Pytorch 2.1。
在实验的超参数上,输入图片长宽均为112,预训练阶段采用Adam优化器,起始学习率为0.0001,采用线性学习率衰减,批尺寸为32,共训练200轮。线性微调阶段同样采用Adam优化器,起始学习率0.01采用余弦退火的学习率衰减策略,批尺寸128,共训练15轮。
3.3. 对⽐实验
MicDiffRep在脑瘤分类数据集上的线性微调准确率结果如表1所示。本文提出的MicDiffRep领先于现有的基于对⽐学习的方法 [5] [6] [7] [8] 和基于图像掩码建模的方法 [9] 。并且,经过MSFA模块对各层特征进行聚合之后,MicdiffRep-MS的准确率提升了多达6个百分点。在现有方法中,基于图像掩码建模的方法MAE [9] 的准确率显著低于其他方法,这是因为在图像掩码建模的预训练过程中,大量的图像区域被掩码覆盖掉,模型很难学习到图像的细节纹理信息,而这些信息对于脑瘤图像的分类是至关重要的。而本文提出的基于扩散模型的MicDiffRep方法则能够尽可能地保持图像的细节纹理信息,并在脑瘤图像分类中达到最优的性能。

Table 1. System resulting data of standard experiment
表1. 在脑瘤分类数据集上的线性分类准确率对比
3.4. 消融实验
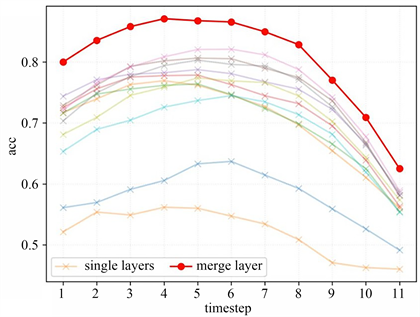
Figure 4. Diagram of the practical teaching system of automation major
图4. 单层特征和聚合特征的线性准确率对比
为了进一步探究在本⽂提出的MicDiffRep方法中,不同的时间步(即不同的噪声程度)和模型的不同层的特征的表征能力,并验证多尺度特征聚合模块MSFA是否有效地提升了模型对于图像全局信息的理解,我们设计了对应的消融实验。分别使用各个不同的单层特征和经过MSFA模块的聚合特征,在不同时间步的特征进行线性微调。图4展示了实验的结果。图中横轴为不同的时间步,纵轴为线性分类的准确率,不同的叉号曲线表示模型的不同单层特征,圆圈曲线表示聚合特征。可以看到,经过MSFA模块的聚合特征的准确率大幅高于各单层特征,这验证了多尺度聚合设计的有效性。观察准确率随时间步的变化,可以看到,在加入适量高斯噪声时,模型产出特征的表征能力最强,而加入过小或过大的噪声时,模型性能都稍差。这是因为,扩散模型预训练阶段,输⼊到模型的图片一般都含有强度适中的高斯噪声。
3.5. 可视化实验
扩散模型最初用于生成图片,本文提出的MicDiffRep表征学习方法也是基于扩散模型能够理解图像的结构和细节纹理特征。因此,生成图片的质量也能从侧面反映模型对于图像纹理特征的理解能力,进而反映出模型对图像的表征能力。如果模型生成的图像结构合理,细节逼真,那么将其特征用于图像分类,自然也达到很高的精度。
为了探究MicDiffRep生成图片的质量,进而验证模型对图像结构和细节的理解能力,我们对预训练阶段扩散模型的生成结果进行了可视化。在生成参数的设置上,我们与标准DDPM [12] 的训练和采样的线性时间步策略保持一致,总的时间步为1000。图5展示了MicDiffRep生成的医学图像与数据集中的真实医学图像。可以看到,本文提出的MicDiffRep生成的医学图像细节逼真,在整张图片的结构上也很合理,整体来看,与真实的医学图像几乎难辨真假。这说明,MicDiffRep在预训练时理解了医学图像的纹理细节和图像结构,从而在进行医学图像分类时,取得了最优的性能。
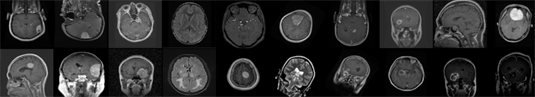

Figure 5. Generated medical Images (upper two rows) and real medical images (bottom two rows)
图5. MicDiffRep生成的医学图像(上方两行)与真实医学图像(下方两行)
4. 总结
本⽂提出了一种基于扩散模型的自监督医学图像分类方法MicDiffRep。在扩散模型预训练时,保持完整的图像全局信息和细节纹理信息,模型得以理解医学图像的图像结构和纹理细节,再通过本文提出的MSFA模块,将各层特征聚合,利用图像的全局信息。综合以上优势,在脑瘤图像分类任务上,MicDiffRep相较于现有最优的自监督表征模型提升多达6个百分点。