1. 引言
真鲷虹彩病毒病(Red sea bream iridoviral disease, RSIVD)属于虹彩病毒科,病毒有囊膜,核衣壳为二十面体,直径200~260 nm,病毒核酸为双链线状DNA [1] 。该病1990年在日本真鲷养殖场首次暴发,主要危害幼鱼,发病后死亡率高达37.9%。发病期在每年7~10月,水温22.6℃~25.5℃为发病最高峰;水温降至18℃以下,可自然停止发病。真鲷虹彩病毒病属于二类传染病。感染真鲷虹彩病毒的症状为病鱼体色变黑,嗜睡,贫血症状明显 [2] ;体表和腮出血,腮上有瘀斑,呈灰白色;脾脏肥大,在光镜下可观察到病鱼的脾脏、肝脏、心脏、肾脏或腮组织中有被Giemsa染色的异常肥大的细胞,内有许多病毒粒子 [3] 。一般可从病鱼体表、鳃的外观症状和脾脏肥大,作出初步诊断。通过取病鱼脾脏、肝脏、心脏、肾脏或鳃组织,切片,Giemsa染色,可在光镜下观察到异常肥大的细胞,也可做肾脏超薄切片,通过电镜观察病毒粒子,或用BF-2、LBF-1等细胞株分离培养病毒,用直接免疫荧光抗体技术检测 [4] 。袁向芬 [5] 等建立环介导等温扩增方法,作为口岸一线的疫病监测。Yasuhiko Kawato [6] 通过eDNA监测确定了养鱼场RSIVD的感染源,并可将其应用于水产养殖中,以控制鱼类疾病。本研究结合实验室检测标准、方法,采用多种分子生物学手段开展检测和鉴定。
2. 材料方法
以刺尾鲷科海水观赏鱼为本次研究材料,样本编号为5147。使用核酸提取试剂盒(达安基因股份有限公司,DA0591)进行病毒基因组核酸提取。具体操作如下:取三尾样本鱼的腮、肝脏、脾脏等腺体组织300 mg,加入1800 μL灭菌ddH2O,用均质处理器充分匀浆研磨,于−80℃室温反复冻融三次,将组织匀浆液以12,000 r/min、4℃条件离心10 min,取上清液200 μL转移无核酸酶污染的离心管中用于核酸提取,加入裂解液200 μL、蛋白酶K 50 μL,72℃孵育10 min。12,000 r/min高速离心1 min,取上清加入250 μL无水乙醇,充分吹打混匀,转入离心柱,离心去废液,加入500 μL抑制物去除液至离心柱,离心去废液,加入500 μL去离子液,反复离心。离心管内即为核酸溶液,保存待用。
真鲷虹彩病毒试剂盒购买于深圳澳东检验检测科技有限公司。
真鲷虹彩病毒病的扩增引物源自Mohr et al. [7] ,由生工生物工程(上海)股份有限公司合成,引物稀释为10 μmol/L工作液备用。
2.1. 荧光定量PCR法检测
用深圳澳东检验检测科技有限公司提供的真鲷虹彩病毒核酸检测试剂盒(PCR荧光探针法、货号:8C143),进行病毒核酸扩增检测。具体操作如下:加入核酸扩增反应液16 μL,真鲷虹彩病毒反应液2 μL和PCR酶混液2 μL,将体系均匀混合,DNA模板每孔5 μL。PCR扩增循环设置如下:95℃预变性3 min;95℃变性5 s,55℃退火/延伸40 s,45个循环,最后根据Ct值和扩增曲线来判定结果为“阴性”或“阳性”。
2.2. SYBR Green法检测
使用诺唯赞Master Mix (诺唯赞,7E461K0)配合前述引物工作液,进行病毒扩增检测。引物由生工生物工程(上海)股份有限公司合成。将提取的DNA核酸(1 µL)加入Taq聚合酶缓冲液中,该缓冲液含有1mM的引物、200 mM的脱氧核苷酸三磷酸和1.25U的EX Taq DNA聚合酶(在20 mM Mg2+ PCR缓冲液中)。PCR扩增循环设置如下:94℃、30 s,58℃、60 s,和72℃、60 s,最后在72℃条件下保持5 min,反应体系30个循环。
2.3. 毛细管电泳仪检测
使用Qsep1毛细管电泳仪(杭州厚泽生物科技有限公司)将上述PCR产物进行毛细管电泳分析。具体操作如下:2 μL PCR产物加入18 μL的disulation buffer充分混合,备用。在前五孔中分别加入200 µL的Separation Buffer、50 µL的纯水、200 µL的纯水、30 µL的Alignment Marker和30 µL的Size Marker,在后三孔分别加入阳性对照和待检测样本(两个重复),离心去气泡,上机读取数据。
2.4. 测序及系统发育树构建
取30 μL的PCR产物和所对应的真鲷虹彩病毒上下游引物,交由生工生物工程(上海)股份有限公司进行测序,测序回来的序列构建进化树。使用MEGA6软件的Neighbor-Joining Tree (NJ邻接统计方法)构建一棵有根的系统发育树 [8] [9] 。采用p-distance方法获得进化距离,并利用进化距离来估算每个位点的氨基酸替换数,通过1000次bootstrap来建立系统发育树的可靠性,自展值 > 50%为可信度 [10] [11] 。以Nervous necrosis virus coat protein mRNA (DQ864760.1)参考,使系统发育树更具有说服力。
3. 结果与分析
3.1. 荧光定量PCR检测出真鲷虹彩病毒
目前荧光定量PCR仪器以荧光强度为y轴、用循环数为x轴,能自动分析得到荧光强度曲线,直接读取检测结果。基线和阈值设定原则根据仪器噪声进行调整,以阈值线刚好超过正常阴性样品扩增曲线的最高点为准。结合表1和图1可知阴性曲线和提取对照曲线无典型的S型扩增曲线且无Ct值;两个阳性曲线的出现典型的S型扩增曲线且Ct值小于30.0,说明质控标准符合要求。根据本样品(5147)数据,Ct值为20.57,且出现明显的指数增长期,表示该样本为阳性检出。
3.2. SYBR Green法检测出真鲷虹彩病毒
荧光定量PCR (SYBR Green)是收集荧光信号强度,通过将荧光强度与循环数作图,qPCR仪生成扩

Figure 1. Amplification curve of red sea bream iridovirus disease
图1. 真鲷虹彩病毒扩增曲线

Table 1. Ct values corresponding to the detection of red sea bream iridovirus disease
表1. 真鲷虹彩病毒检出所对应的Ct值
增曲线,表示在整个PCR过程中产物的累积,通过这个过程,即可实现量化。由图2所知在早期循环中,荧光信号没有变化,随着反应的进行,每个循环的荧光水平开始显著上升,出现典型的指数增长期。从图中的Ct值及曲线模型观察,样品Ct值21.16,且与阳性样品一样出现典型的S型扩增曲线,初步判定为阳性检出。以荧光信号随时间变化作图和以荧光变化/温度变化与温度作图,可得到熔解曲线图,特异性扩增的熔解曲线是一个紧密的单峰,该图谱表明样品的曲线与阳性的曲线几乎重合且是紧密的单峰,说明样品为阳性检出。
3.3. 毛细管电泳仪检测出真鲷虹彩病毒
便携式毛细管电泳检测能高效、快速、精准检测出样本片段大小。结果如图3所示,我们同时测定了阳性和5147两个样本,图3(a)图为合成质粒阳性,电泳测出其片段为565 bp,其值在合理有效范围内Alignment Marker (20~1 K)且在该范围内杂峰较少,说明引物特异性好,阳性质粒浓度较高;图3(b)图为5147样本峰图,电泳检测出片段大小为581 bp,该片段在Alignment Marker (20~1 K)范围值内,5147样本没有杂峰。真鲷虹彩病毒特异性片段大小为570 bp,根据毛细管电泳仪说明其值在±10%范围内浮动属于合理有效范围,由此可知5147样本为真鲷虹彩阳性样本检出。图3(d)为补充说明,两个样本均在有效范围内且无杂峰,证明为阳性检出。
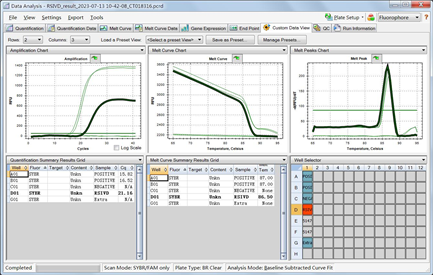
Figure 2. Amplification curve of fluorescent PCR for red sea bream iridovirus disease
图2. 真鲷虹彩病毒荧光PCR扩增曲线
3.4. 序列比对及系统发育树构建
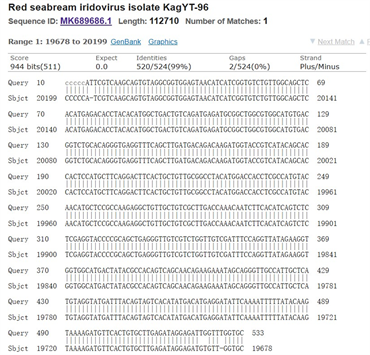
获得的序列在NCBI上比对完毕,我们选取得分率最高的数据。如图4(a)所示E值代表随机匹配的可能性,E值越小,序列越匹配。E值接近于0,基本说明完全匹配上。其中比对上的序列长度为533 bp,笔者提供的序列长度为524 bp,其中520 bp匹配上。由序列比对得出,受测样本确定为真鲷虹彩病毒。该分析涉及12个核苷酸序列,如图4(b)所示,通过对5147样本的序列比对,构建了系统发育树。系统发育树表明12个核苷酸序列中,有11份序列比对样品为真鲷虹彩病毒分离株,另一份序列为外源参考序列,序列比对和系统发育树表明5147为阳性检出。
 (a)
(a)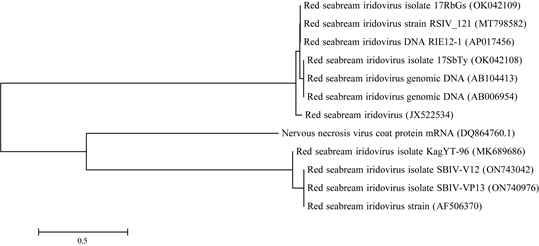 (b)注:括号内的数字为GeneBank登录号;图上方标尺数字代表遗传距离,线段距离越长代表亲缘关系越远。
(b)注:括号内的数字为GeneBank登录号;图上方标尺数字代表遗传距离,线段距离越长代表亲缘关系越远。
Figure 4. Constructs a phylogenetic evolutionary tree based on gene sequence alignment
图4. 基于基因序列比对构建系统发育进化树
4. 结论
真鲷虹彩病毒是《中华人民共和国进境动物检疫疫病名录》规定的二类动物疫病,是进境水生动物疫病重点监测项目。真鲷虹彩病毒能引起大黄鱼、石斑鱼等死亡,给水产养殖业造成重大经济损失,严重影响了水产养殖业的健康发展。建立真鲷虹彩病毒的SYBR Green荧光定量PCR法,SYBR染料法需要注意引物的特异性,因为非特异扩增产物和引物二具体均为dsDNA,所以SYBR染料法只能通过引物来保证特异性,但也可借用熔解曲线初步判断,避免进行常规凝胶电泳SYBR染料法的优点在于简单且成本较低,对荧光探针标记特定的核苷酸片段进行定性检测;荧光探针法试剂盒检测方法灵敏准确、操作简单,但可能存在设计缺陷,不利于进行后续电泳及测序检测;利用毛细管电泳,高分辨率卡夹,多重检测,对获得的PCR产物进行核酸片段大小测定,毛细管电泳判断片段大小是否均一、符合预期具有操作简单,所需时间短、结果直观等优点,但不可避免存在仪器贵、耗材成本高等缺点;测序分析比对靶向针对性好,结果参考价值高,但PCR扩增产物送测存在扩增错配延伸的可能,且操作较为复杂、测序产物纯化难度大。再利用NCBI数据库进行序列比对,构建系统发育树,丰富真鲷虹彩病毒的基因组信息库、分子溯源数据库,为病原微生物的归类鉴定提供准确数据。这些检测方法在鱼类养殖业中的应用,可以帮助养殖户及时发现和控制病毒的传播,减少损失并保障鱼类的健康。
基金项目
汕头海关科研项目(2022STK004;2022STK005)。
NOTES
*第一作者。
#通讯作者。