1. 引言
腹主动脉瘤的起始是由腹主动脉中间层的退化改变所引起的。这样的改变导致了动脉瘤的生成,这一过程伴随着在血压的影响下动脉层的扩张,最终导致动脉壁永久性地增大,其扩张程度超出了正常血管直径的150% [1] 。腹主动脉瘤破裂后的病死率将超过80%,由此成为最具威胁性的心血管疾患之一。腹主动脉瘤的临床表现为腹部主动脉壁局部的异常扩大 [2] 。腹主动脉瘤(AAA)每年在全球导致约17万人死亡。大多数指南建议,无症状的小型AAA (女性为30至<50毫米;男性为30至<55毫米)通过影像学监测,而大型无症状的、有症状的及破裂的AAA应考虑进行外科修复 [3] 。当前的术后结果仍然令人关注,因为根据现代公开发表的系列研究,发病率和死亡率仍然持续在25%至50%的范围内 [4] 。腹主动脉瘤可以通过药物或手术进行治疗,具体取决于患者的症状以及动脉瘤的大小和生长速度。药物治疗适用于无症状患者和较小的动脉瘤,当男性动脉瘤直径大于5.5 cm,女性动脉瘤直径大于5.0 cm时,应进行手术治疗,包括开放性动脉瘤和血管内动脉瘤修复术 [5] 。因此,识别促成腹主动脉瘤发病与进展的新型分子靶点具有极高的临床价值。生物信息学分析已成为医学领域科学研究的重要技术手段 [6] 。数据集的整合及评估能够减少小规模实验研究中的不准确性,增强研究结果的可靠性。微阵列平台以其高通量分析能力,已成为发现疾病状态下遗传学或表观遗传学重要变化的有力而有效的方法,它是识别用于病症诊断和预测的潜在生物标记物的宝贵资源 [7] 。在过去的研究表明,T细胞、巨噬细胞、树突状细胞、中性粒细胞、B细胞和肥大细胞的多种炎症细胞浸润进入主动脉壁,它们在腹主动脉瘤发展中起着至关重要的作用 [8] 。炎症在腹主动脉瘤(AAA)发病机制中的重要性已被证实,而且在炎症中增加的氧化应激同样在炎症的病理生理过程中扮演着至关重要的角色 [9] 。在炎症反应中,氧化应激由活性氧种(ROS)的增加通过多种机制促进炎症,这些机制包括但不限于激活转录因子如NF-κB (核因子κB)、促进炎症细胞因子的释放、损伤组织结构以及激活信号通路 [10] 。此外,内皮细胞、平滑肌细胞和成纤维细胞也会产生蛋白酶,这些蛋白酶分解内弹性层,进而加剧腹主动脉瘤(AAA)的进展。包括成纤维细胞、肌成纤维细胞和“合成型”血管平滑肌细胞在内的外膜间充质细胞会释放I型胶原、转化生长因子(TGF)-β和基质金属蛋白酶,这些物质控制细胞外基质的重塑 [11] 。由于病因不明确,除了外科干预手术外,目前临床实践中还没有特定有效的药物治疗方法来预防腹主动脉瘤的发展和破裂。因此,识别与AAA发病和进展相关的新型分子靶点具有重大的临床意义。基因表达综合数据库(Gene Expression Omnibus, GEO)成为了最全面的公开基因表达数据资源库 [12] 。
2. 方法
2.1. 数据集的基因差异表达分析
在GEO中检索与AAA相关的数据芯片。通过共享处理平台GPL10558 (Illumina HumanHT-12 V4.0表达芯片)提取与AAA相关的数据芯片GSE47472、GSE57691和GSE119717。这些数据芯片均由同一团队依次上传,并通过相同的平台检测(表1)。这确保了源样本的一致性和测序过程的一致性。我们分析了腹主动脉瘤的样本(共14个,平均最大主动脉直径为62.6 ± 18.0毫米)的差异表达基因。这些样本与GSE47472数据集中的对照主动脉供体标本(共8个)进行了比较。每个样本均来自完整厚度的主动脉壁,这减少了样本位置差异对最终分析结果的影响。进一步分析了GSE57691数据集中20例小型腹主动脉瘤患者(平均最大主动脉直径54.3 ± 2.3毫米)、29例大型腹主动脉瘤患者(平均最大主动脉直径68.4 ± 14.3毫米)和10个器官捐赠者的对照主动脉标本之间的差异基因表达。同样,我们在GSE119717数据集中探究了30个血管周围增生脂肪组织样本与30个非增生血管周围脂肪组织样本中的差异基因表达。我们使用R语言中的“sva”包来合并,并标准化GSE47472和GSE57691数据集。通过R中的“limma”包(版本4.2.1),我们识别出差异表达基因,将p < 0.05且|log2Fold change (logFC)| > 1大于1视为统计学上具有意义。
2.2. 差异表达基因功能与信号通路富集分析
通过应用基因本体论(Gene Ontology, GO),我们研究了差异表达基因的生物学功能,确定了相关主要细胞组分、分子功能和生物过程。我们评估了京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)途径,以了解与感兴趣基因相关的信号传导途径。这一分析是通过R4.2.1软件中的“clusterProfiler [4.4.4]”程序进行。我们证明了生物过程和途径术语对于差异表达基因(DEGs)是统计学上显著的。考虑观察到的差异,我们确定P < 0.05是根据累积超几何分布检验在统计学上具有显著性。此外,我们提出了基因集富集分析(Gene Set Enrichment Analysis, GSEA),与单基因审查不同,GSEA优先考虑基因集,即共享生物学功能、调控或染色体位置的基因群,并利用这些集合的累积强度 [13] 。通过R (版本4.2.1)中的clusterProfiler R包(版本4.4.4)进行。基于统计分析,p.adj < 0.05和FDR (q值) < 0.25被认为是显著差异。
2.3. 检测功能模块中的核心基因并研究关键基因
我们使用Cytoscape软件构建蛋白质–蛋白质相互作用网络(Protein-Protein Interaction, PPI)。我们利用STRING数据库(蛋白质–蛋白质相互作用网络的功能富集分析)来识别腹主动脉瘤组和对照组中差异表达基因的PPI配对。我们使用STRING数据库以0.40的置信度阈值分析蛋白质相互作用,然后使用R中的igraph包[1.4.1] (版本4.2.1)清洗和整理数据。使用Cytoscape软件构建PPI网络,并使用cytoHubba模块进一步筛选出前10个关键基因。
3. 结果
分析GSE47472、GSE57691和GSE119717数据集(表1),我们根据p < 0.05和|log2Fold change (logFC)| < 1的标准,识别出了819、237和24个差异表达基因。我们将再次使用limma包中的normalizeBetweenArrays函数对数据进行标准化,利用箱式图检查样品情况,通过limma包进行两组的差异评估,使用R中的ggplot2 [3.3.6]通过火山图可视化差异评估结果,并使用R中的Heatmap [2.13.1]以热图格式可视化显著表达的分子(图1、图2和图3)。
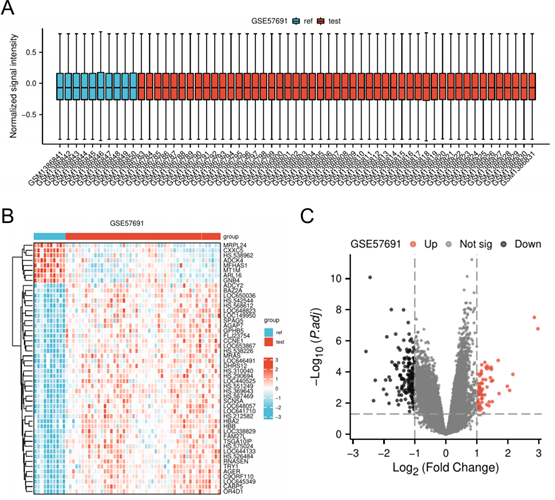 (A) 该箱形图代表了实验组50个样本(红色)和对照组10个样本(蓝色)的标准化数据。箱体内部的水平线代表标准化数据的中位数,箱体的顶边代表上四分位数,底边代表下四分位数。(B) 237个DEGs的热图。该图展示了所有DEGs和样本的双向层次聚类结果。热图中的每一行代表一个样本,每一列代表一个基因。热图右侧的颜色刻度表示原始Z分数,范围从蓝色(低表达)到红色(高表达)。(C) DEGs的火山图。右侧的红色节点代表上调的DEGs,左边的黑色节点代表下调的DEGs。识别差异的标准为:|logFC| > 1且P < 0.05。
(A) 该箱形图代表了实验组50个样本(红色)和对照组10个样本(蓝色)的标准化数据。箱体内部的水平线代表标准化数据的中位数,箱体的顶边代表上四分位数,底边代表下四分位数。(B) 237个DEGs的热图。该图展示了所有DEGs和样本的双向层次聚类结果。热图中的每一行代表一个样本,每一列代表一个基因。热图右侧的颜色刻度表示原始Z分数,范围从蓝色(低表达)到红色(高表达)。(C) DEGs的火山图。右侧的红色节点代表上调的DEGs,左边的黑色节点代表下调的DEGs。识别差异的标准为:|logFC| > 1且P < 0.05。
Figure 1. Differentially expressed genes (DEG) analysis of GSE57691
图1. GSE57691的差异表达基因(DEG)分析
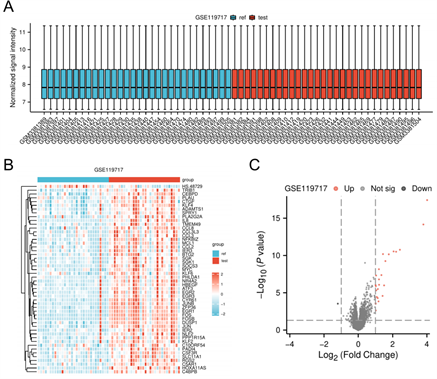 (A) 该箱形图代表了实验组30个样本(红色)和对照组30个样本(蓝色)的标准化数据。(B) 25个DEGs的热图。该图展示了所有DEGs和样本的双向层次聚类结果。(C) DEGs的火山图。右侧的红色节点代表上调的DEGs,左侧的黑色节点代表下调的DEGs。鉴定差异的标准为:|logFC| > 1且P < 0.05。
(A) 该箱形图代表了实验组30个样本(红色)和对照组30个样本(蓝色)的标准化数据。(B) 25个DEGs的热图。该图展示了所有DEGs和样本的双向层次聚类结果。(C) DEGs的火山图。右侧的红色节点代表上调的DEGs,左侧的黑色节点代表下调的DEGs。鉴定差异的标准为:|logFC| > 1且P < 0.05。
Figure 2. Differentially expressed genes (DEG) analysis of GSE119717
图2. GSE119717的差异表达基因(DEG)分析
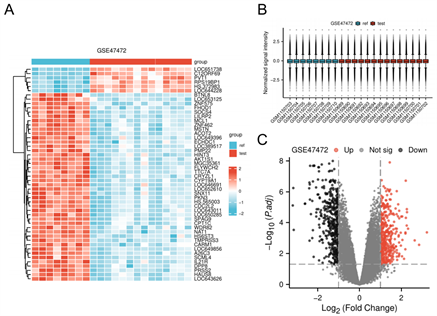 (A) 该箱形图代表了实验组14个样本(红色)和对照组8个样本(蓝色)的标准化数据。(B) 25个DEGs的热图。(C) DEGs的火山图。鉴定差异的标准为:|logFC| > 1且P < 0.05。
(A) 该箱形图代表了实验组14个样本(红色)和对照组8个样本(蓝色)的标准化数据。(B) 25个DEGs的热图。(C) DEGs的火山图。鉴定差异的标准为:|logFC| > 1且P < 0.05。
Figure 3. Differentially expressed genes (DEG) analysis of GSE47472
图3. GSE47472的差异表达基因(DEG)分析
3.1. 差异表达基因的功能与途径富集分析
合并GSE47472和GSE57691的差异表达基因,并通过clusterProfiler [4.4.4] R包分析了1056个差异表达基因的mRNA功能,识别GO和KEGG途径。对455个上调差异表达基因(DEGs)的富集分析显示,生物过程(BPs)中排名前四涉及的生物过程包括对外界刺激的反应、防御反应的上调、一氧化氮代谢过程的调控以及急性炎症反应的增强。细胞成分组中包括富含ficolin-1的颗粒膜、血红蛋白–血红素复合体和横切质膜成分。腹主动脉瘤(AAA)中的分子功能主要涉及血红素结合、细胞因子结合和氧携带分子的活动。KEGG表明,表达活性增高的基因主要参与诸如人类巨细胞病毒感染、趋化因子信号、非酒精性脂肪肝炎以及IL-17信号途径等多种疾病和反应(图4(A))。对601个活性较低的差异表达基因的深入分析
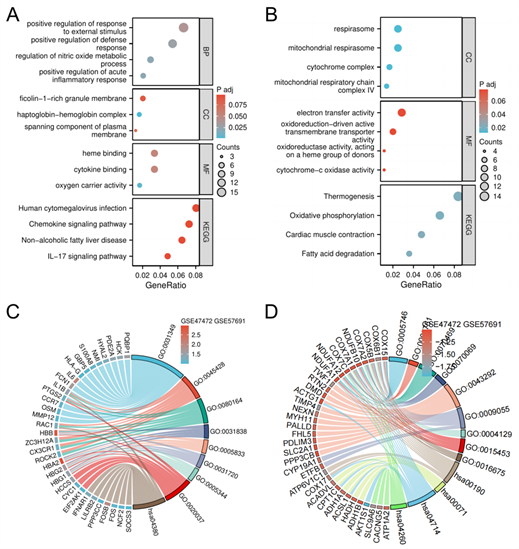
Figure 4. After merging the GSE47472 and GSE57691 data sets, GO and KEGG analyzes were performed on the up- and down-regulated DEGs to compare the differences between the experimental group and the control group. (A) Bubble plot showing the upregulated genes in GO and KEGG analysis. (B) Bubble plot showing downregulated genes in GO and KEGG analysis. (C) Chord plot showing enrichment terms for up-regulated genes. (D) Chord plot showing enrichment terms for down-regulated genes
图4. 合并GSE47472和GSE57691数据集后,对上调和下调DEGs进行了GO和KEGG分析,以比较实验组与对照组之间的差异。(A)展示了GO和KEGG分析中上调基因的气泡图。(B)展示了GO和KEGG分析中下调基因的气泡图。(C)展示了上调基因富集项的弦图。(D)展示了下调基因富集项的弦图
表明,在细胞结构方面的前三项特征包括呼吸体、线粒体呼吸体、细胞色素复合体和线粒体呼吸链的复合体IV。AAA中的分子功能主要集中在活性跨膜转运蛋白的氧化还原诱导激活、作用于供体血红素组上的氧化还原酶活性以及细胞色素c氧化酶的活动。KEGG途径发现下调差异表达基因主要参与热发生、氧化磷酸化、心肌收缩和脂肪酸降解(图4(B))。GSE119717数据集中的差异表达基因进行了验证,通过R中clusterProfiler [4.4.4]包进行的GO和KEGG途径分析,鉴定出24个上调基因。生物过程涵盖细胞趋化性、对细菌源分子的反应、粒细胞趋化性以及嗜中性粒细胞趋化性。细胞结构还包括诸如富含胶原的细胞外基质、内质网内部和膜附着元素等成分。AAA的分子功能主要集中在特定DNA结合转录激活因子活性、与RNA聚合酶II有关的细胞外基质成分以及G蛋白偶联受体结合。上调差异表达基因的KEGG途径结果表明,mRNA主要参与PI3K-Akt信号途径、IL-17信号途径、TNF信号途径以及病毒蛋白与细胞因子及细胞因子受体的相互作用(图5(A))。
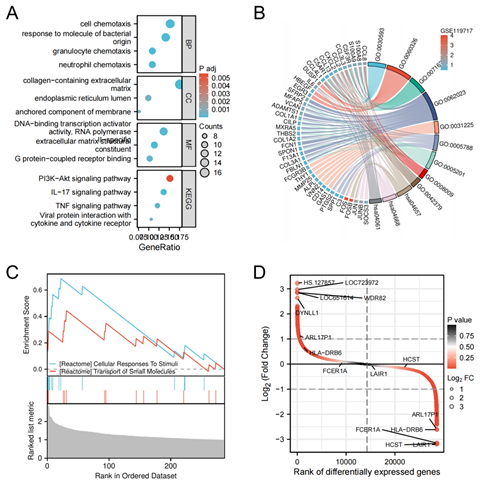
Figure 5. Bubble chart (A) showing the enrichment analysis of up-regulated differentially expressed genes in the GSE119717 data set. Chord plot (B) showing enrichment analysis. GSEA enrichment analysis of differentially expressed genes after merging GSE47472 and GSE57691 datasets is shown. The criteria for identifying differences are: p.adj < 0.05 and FDR (q value) < 0.25. (C) The difference ranking diagram shows the gene differential expression ranking obtained by merging the GSE47472 and GSE57691 data sets, in which |LogFC| > 1 and Threshold of p < 0.05. The horizontal axis indicates the ranking position of logFC, while the vertical axis represents logFC. The top five and bottom five genes are marked (D)
图5. 展示了GSE119717数据集中上调差异表达基因富集分析的气泡图(A)。展示了富集分析的弦图(B)。显示了合并GSE47472和GSE57691数据集后差异表达基因的GSEA富集分析。鉴定差异的标准为:p.adj < 0.05且 FDR(q值) < 0.25。(C) 差异排名图展现了通过合并GSE47472和GSE57691数据集获得的基因差异表达排序,其中应用了|LogFC| > 1和p < 0.05的阈值。水平轴指示logFC的排名位置,而垂直轴表示logFC。标注了排名前五和后五的基因(D)
3.2. 基因集富集分析
基因本体(GO)用于识别由差异基因表达引起的功能性变化,而京都基因与基因组百科全书(KEGG)用于识别途径级别的变化。在GO和KEGG中进行差异基因的富集评估需要定义阈值,这可能会排除生物学上重要的基因。基因集富集分析(GSEA)不需要明确的差异基因阈值,并基于实际的全局趋势进行分析;因此,其结果可以补充GO和KEGG的富集评估。在差异分类图中,标记了排名前五的上调和下调基因。(图5(D))我们使用ggplot2[3.3.6]和R的clusterProfiler[4.4.4]对数据集中选定的差异表达基因进行GSEA富集分析和可视化(图5(C))。差异的识别基于以下标准:p.adj < 0.05且FDR(q值) < 0.25。由于GSE119717中差异表达转录本有限,因此未进行GSEA富集分析。
3.3. 构建蛋白质–蛋白质相互作用(PPI)网络和分析关键基因
STRING是一个在线存储库和工具,用于预测和证实蛋白质–蛋白质相互作用作为生物数据集,接纳了所有的差异表达基因(DEGs)。通过Cytoscape软件中实验、邻域、数据集、文本挖掘和共表达的参数构建PPI网络后,孤立的基因被消除。图6(A)显示了合并的GSE47472和GSE57691数据集中具有差异表达的基因之间的相互作用,图6(B)显示了GSE119717数据集中具有差异表达的基因之间的相互作用。CXCL8,SOCS3,IL6,PTGS2,EGR1,FOS,ATF3,DUSP1,JUN,MY (图6(C));CXCL8,SOCS3,IL6,PTGS2,FOS,OSM,IL1B,S100A8,HCK,NCF2 (图6(D))。交集基因包括SOCS3、IL6、PTGS2、FOS (表2)所有这些关键基因都可能具有临床相关性。
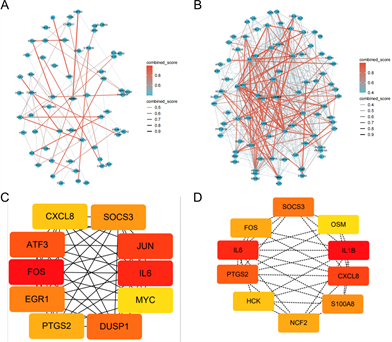
Figure 6. PPI analysis of candidate differentially expressed genes, and the interrelationships between candidate genes screened in the merged GSE47472 and GSE57691 data sets are shown in the network diagram (A). The interrelationships between candidate genes screened in the GSE119717 data set are shown in the network diagram (B). PPI subnetwork of the top 10 central candidate genes merging the GSE47472 and GSE57691 datasets (C). PPI subnetwork (D) of the top 10 central candidate genes of the GSE119717 dataset. PPI refers to protein-protein interaction
图6. 候选差异表达基因的PPI分析,在合并后的GSE47472和GSE57691数据集中筛选的候选基因之间的相互关系在网络图中展示(A)。在GSE119717数据集中筛选的候选基因之间的相互关系在网络图中展示(B)。合并GSE47472和GSE57691数据集的前10个中心候选基因的PPI子网络(C)。GSE119717数据集的前10个中心候选基因的PPI子网络(D)。PPI指的是蛋白质–蛋白质相互作用
AAA: abdominal aortic aneurysm.
3.4. 腹主动脉瘤关键基因的临床意义
敏感性与特异性的关系由ROC曲线表示。图7展示了一个ROC曲线,表明4个关键基因对腹主动脉瘤(AAA)的诊断敏感性出色。其中,FOS的AUC值为0.963,表明这一基因可能成为诊断AAA的潜在生物标记物。

Figure 7. Four selected genes were evaluated using ROC curves. The area under the ROC curve (AUC) is often used to evaluate the performance of a diagnostic test. AUC ranges between 0.5 and 1, with higher AUC indicating better diagnostic accuracy in predicting outcomes. The closer the AUC is to 1, the better the variable predicts the outcome
图7. 利用ROC曲线对四个选定基因进行了评估。ROC曲线下面积(AUC)通常用以评估诊断测试的效能。AUC的范围在0.5到1之间,较高的AUC表明在预测结果方面具有更好的诊断准确性。AUC越接近1,说明变量对结果的预测性能越好
4. 讨论
腹主动脉瘤(AAA)指的是腹主动脉持续性扩张超过3厘米直径的情况。大多数AAA病例发现在肾下段,即肾下型腹主动脉瘤 [14] 。本研究利用生物信息学方法从基因表达综合数据库中提取、分类和筛选有关人类AAA基因的表达信息。利用生物信息学研究健康个体(对照组)与AAA患者的主动脉壁样本之间差异表达的潜在基因。然后利用基因富集分析评估生物学相关性。SOCS3、IL6、PTGS2和FOS被识别为与AAA的发病或治疗相关的关键差异表达基因(DEGs)。这三个DEGs可能作为重要的生物标记物,为AAA的发病机制和进展提供机理见解。
目前,AAA的进展被认为与炎症、氧化应激、平滑肌细胞的凋亡以及细胞外基质的降解有关 [15] 。血管损伤经常与大多数心血管疾病中的炎症相关,可以导致内皮细胞死亡和血管壁结构破坏 [16] 。随着免疫反应的激活,AAA壁发生蛋白水解性细胞外基质的破碎以及平滑肌细胞(VSMC)表型的改变或凋亡,导致显著的中层变薄和降解,VSMC数量减少,以及在动脉瘤壁中弹性元素的减少 [17] [18] 。通过分析GSE47472、GSE57691和GSE119717数据集中的差异表达基因,确定了479个上调和601个下调基因。这些差异表达基因(DEGs)进行了途径富集分析。前五个富集的GO包括积极调节对外界刺激的反应、防御反应、一氧化氮代谢过程、急性炎症反应和细胞外基质结构成分。TNF信号通路、人类巨细胞病毒感染、趋化因子信号通路、非酒精性脂肪肝病和IL-17信号通路是五个最富集的KEGG途径。CXCL8、SOCS3、IL6、PTGS2和FOS被认为是AAA形成中的重要差异表达基因。根据近期研究,FOS在AAA的发展中起着关键作用 [19] 。FOS基因表达的增加通过增强炎症反应,促进了腹主动脉瘤(AAA)的发展。此外,FOS基因表达也与主动脉壁的破坏和肿瘤内血管生成有关,为其在AAA发展中的作用提供了额外的证据。考虑到其促进作用,FOS基因可能为AAA的治疗提供了新的潜在方法和靶点 [20] 。一项前瞻性研究发现FOS基因与AAA发展中的氧化应激途径有显著关联。总体而言,FOS基因是AAA发展的关键因素 [21] 。在AAA的发起和进展中,腹主动脉外膜中的单核细胞在成纤维细胞的刺激下分泌IL6。这种释放促进了它们向巨噬细胞的分化并加剧了巨噬细胞的激活 [22] 。SOCS3基因在多种疾病的发生和进展中的功能不仅仅是负向调节细胞信号通路。最近的研究已经展示了它在腹主动脉瘤(AAA)中的核心作用 [23] 。SOCS3基因通过负向调节JAK/STAT信号通路影响AAA的发展,该通路调控炎症和凋亡。在AAA患者中,SOCS3基因的表达显著下降,且其表达水平与AAA的直径显著负相关 [24] 。基于这些发现,SOCS3基因参与了AAA的发病和进展,且在炎症和凋亡途径中发挥了关键作用 [25] 。此外,一些研究发现,长期的炎症可以导致SOCS3基因的DNA甲基化,从而引起基因的下调 [26] 。AAA患者中PTGS2基因的表达水平显著上升,并与AAA的大小正相关。PTGS2基因的活性和表达水平与AAA的炎症反应和凋亡过程有着密切的关联。与先前的研究 [27] [28] 相符,这些见解强调了PTGS2基因在AAA的发病机制中的核心角色。由PTGS2基因编码的COX-2酶在前列腺素的生物合成中具有重要作用——前列腺素是参与各种生理和病理过程的生物活性化合物。
我们的研究存在一些局限性。这项研究确定了DEGs在AAA中的表达强度,但没有深入探讨这些潜在基因在动物模型或AAA细胞中的具体机制。进行额外的研究以进一步探索这些机制是必要的。
5. 结论
总结而言,我们的生物信息学分析识别出15个潜在基因,包括SOCS3、IL6、PTGS2和FOS这四个关键基因,这些基因可能与AAA的发病机制和发展有关。这些洞见增强了我们对AAA的理解,并提供了预防、治疗和指导。
NOTES
*通讯作者。