1. 引言
脂肪氧合酶是一种能催化氧化多不饱和脂肪酸(亚油酸、亚麻酸、花生四烯酸)及其酯的普遍存在于真菌、动植物界的酶,其催化过程的产物是氢过氧化脂肪酸或酯 [1] 。脂氧合酶是机体的一部分,它对促进炎症的反应起着关键的作用,而炎症是一种与癌症、脑卒中、心血管及神经退化有关的病症 [2] [3] 。脂氧合酶在植物贮存过程中会产生能改变食品特性的芳香化合物,进而可能会引起食物的变质 [4] [5] 。
亚油酸是脂氧酶最为理想的底物,主要的催化机理也是基于亚油酸提出的,并且获得了优化的反应条件和满意的产率。对亚油酸以外的可识别底物(如甘油酯、磷酸酯和油酸等),研究只是证实了LOX催化它们的可行性,多作为动植物体内的一种代谢路线进行研究。至于脂氧酶对这些底物的催化活性和最终产率,则少有研究或结果很不理想 [6] [7] 。脂氧酶与这些底物分子的结合模式和催化机理尚不明确,这些限制了脂氧酶催化机理的进一步阐明和更好的利用 [8] [9] 。
Discovery Studio是一款综合应用于生命科学预测领域的软件,集成了目前分子模拟领域的各种经典、先进算法及高质量的图形界面。在DS软件中,最适合于酶这类生物大分子与底物小分子进行分子对接的为CDOCKER模块 [10] [11] [12] 。CDOCKER模块是基于CHARMm的柔性对接程序,利用软核和选择性重定向技术,实现配体与受体之间的连接。先将小分子结构采用分子动力学的方法进行随机搜索,再在受体的活性位置对小分子的结构构象进行处理,这样使得分子对接的结果越加精确。该软件的View Interaction模块可以查看底物分子与脂氧酶蛋白质受体的相互作用方式。
本文选择大豆脂氧酶LOX-1为研究模板。LOX-1不仅纯度高,而且热稳定性相对较好。脂肪氧合酶LOX-1的3D结构如图1所示。选择亚油酸、亚油酸甲酯为底物,考察两者与脂氧酶反应的活性差异。利用计算模拟技术将亚油酸、亚油酸甲酯与大豆脂氧酶LOX-1进行分子对接,得到对接之后的结合能等变化的具体数据,观察甲酯基的引入对底物分子与脂氧酶蛋白质受体的相互作用方式的改变,为脂氧酶与底物分子的结合模式研究积累数据。
2. 实验部分
2.1. 实验试剂与仪器
大豆脂肪氧合酶LOX-1 (70,600 U/mg, Sigma);亚油酸(苏州乳化剂厂,66.7%),亚油酸乙酯(99%);二甲酚橙(AR,上海试剂三厂);过氧化氢异丙苯(80%, Sigma);硼酸,硼砂,氢氧化钠,2,6-叔丁基-4-甲基酚(BHT),邻苯二甲酸氢钾(基准试剂),浓硫酸(98%),乙醚,无水乙醇,甲醇,六水合硫酸亚铁铵,均为AR级(国药集团化学试剂有限公司),紫外可见分光光度计(TU-1901,北京普析通用)。
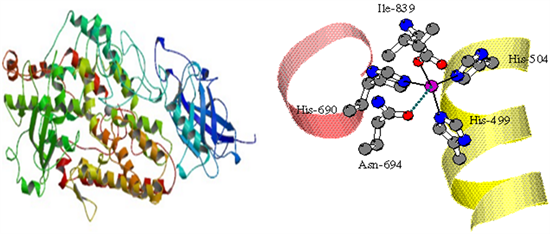
Figure 1. Schematic diagram of the three-dimensional structure and active centers of soybean lipoxygenase-1 (PDBID: 1YGE)
图1. 大豆脂肪氧合酶LOX-1的三维结构和活性中心示意图(PDBID: 1YGE)
2.2. 实验方法
2.2.1. 脂氧酶酶活测定
LOX活性在234 nm处用分光光度法测定。根据Surrey方法制备底物亚油酸溶液 [13] 。酶活定义为在25℃,pH9.0条件下,以亚油酸为底物,3 mL反应体系每分钟在234 nm处增加的吸光度0.001对应一个酶活单位。
2.2.2. 脂氧酶好氧催化反应
在气/液反应器中以0.5 mL乙醇和pH9的磷酸缓冲液稀释1.5 g亚油酸,于15℃恒温水浴中预热5 min后,通入15 mL/min氧气5 min,再加入8 mL酶活为1.5 × 105的LOX-1溶液(溶解于0.1 M pH9的硼砂缓冲液)。反应120 min后用1 mol/L硫酸调节pH至3,用乙醚萃取三次得粗产物。空白样品(自动氧化)是在相同的条件下,利用不含底物的脂氧酶溶液进行反应。每个转化反应重复进行两次,结果取两个独立实验的平均值。考察pH6下的脂氧酶催化反应时,采用0.1 M pH6的磷酸缓冲液,其余条件不变。采用二甲酚橙法测定氢过氧化亚油酸和氢过氧化亚油酸甲酯的含量 [14] 。
2.2.3. 分子对接
从PubChem (https://pubchem.ncbi.nlm.nih.gov/)上下载得到底物亚油酸和亚油酸甲酯的SDF结构式。用Discovery Studio对亚油酸、亚油酸甲酯进行补全H原子和施加力场等优化。通过PDB蛋白质数据库得到脂氧酶1YGE的三维结构数据,去除蛋白质当中的结晶水,自动补全缺失的H。确定最终活性位点并利用CDOCKER模块对底物小分子和脂氧酶进行对接,获得对接数据。实验记录CDOCKER-ENERGY、是否存在氢键作用以及氢键的键长和键角。采用3D条带图和2D显示图将对接结果可视化。
3. 实验结果
3.1. 脂氧酶LOX-1催化亚油酸及其甲酯的反应
以亚油酸为底物时脂氧酶LOX-1催化反应的最佳pH范围为9~10。而亚油酸酯为底物的情况下,LOX-1的最佳反应pH为6~7。说明pH的改变导致酶蛋白的微环境发生了改变,致使LOX催化行为也发生了变化。体系pH值会影响酶的构象以及解离状态,酸性条件下LOX的构象更有利于与脂肪酸酯类底物的结合 [8] 。本实验在pH 6和pH 9的条件下考察了脂氧酶催化亚油酸、亚油酸甲酯的反应,结果如图2所示。pH 9时氢过氧化亚油酸的产率为58.5%,而氢过氧化亚油酸甲酯的产率为14.2%。pH 6时氢过氧化亚油酸的产率大幅下降为28.6%,而氢过氧化亚油酸甲酯的产率略升高为16.8%。亚油酸较亚油酸甲酯与LOX-1的反应活性更强,推测其与LOX-1络合成的反应中间体更为稳定。
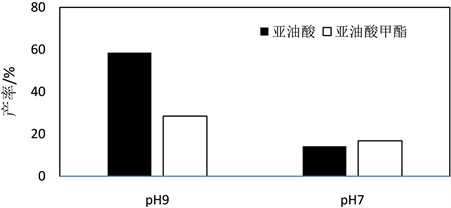
Figure 2. Lipoxygenase-1 catalyzes the reaction of linoleic acid and methyl linoleic acid
图2. 脂氧酶LOX-1催化亚油酸、亚油酸甲酯的反应
3.2. 亚油酸与脂氧酶LOX-1的分子对接图
使用Discovery Studio 2019软件中的CDOCKER模块完成分子对接。首先将亚油酸载入到DS软件中,将大豆脂氧酶蛋白质LOX-1定义为受体分子,利用Tools模块寻找受体中的结合位点。通过View Interaction模块查看亚油酸小分子与蛋白质受体LOX-1的相互作用方式。
亚油酸与大豆脂氧酶LOX-1的分子对接结果如图3 (3D条带显示图)和图4 (2D作用图)所示。绿色部分为参与范德华力作用的氨基酸,亚油酸与脂氧酶LOX-1的VAL354,GLN697,HIS499,LEU546,HIS504和TRP500等22个氨基酸残基有范德华相互作用。SER491和GLN697参与极性作用,ARG707和HIS494参与电荷作用。LOX-1和亚油酸复合物的稳定性由上述这些作用力共同维系。该结果与之前文献报道基本相符 [7] 。
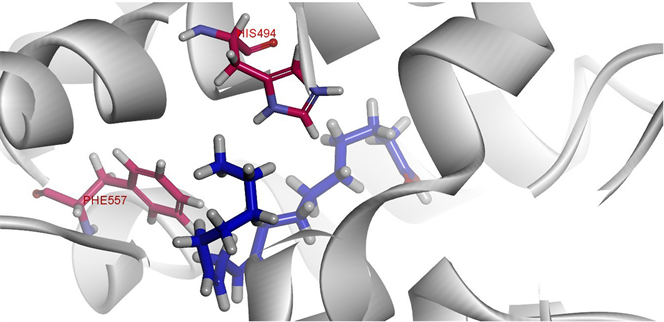 红色结构模型为脂氧合酶上的氨基酸残基,由红色字体标注出氨基酸残基名称;蓝绿色部分为脂氧合酶大分子模型;蓝色部分为亚油酸结构模型。
红色结构模型为脂氧合酶上的氨基酸残基,由红色字体标注出氨基酸残基名称;蓝绿色部分为脂氧合酶大分子模型;蓝色部分为亚油酸结构模型。
Figure 3. Structural model of soybean lipoxygenase-1 and linoleic acid complex
图3. 大豆脂氧合酶LOX-1和亚油酸复合物的结构模型
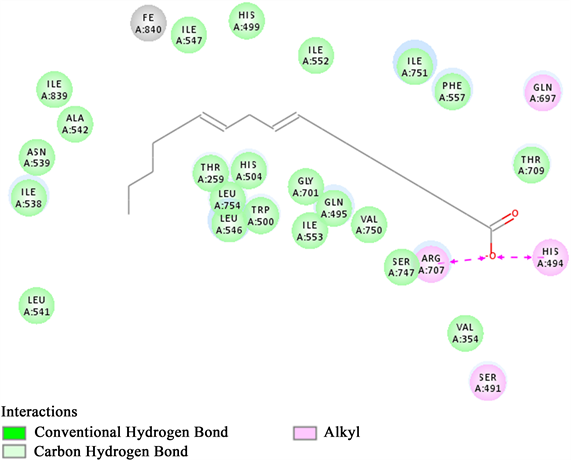
Figure 4. 2D docking diagram of soybean lipoxygenase-1 and linoleic acid
图4. 大豆脂氧酶LOX-1和亚油酸的2D对接作用图
3.3. 亚油酸甲酯与脂氧酶LOX-1的分子对接图
亚油酸甲酯与大豆脂氧酶LOX-1的分子对接结果如图5 (3D条带显示图)和图6 (2D作用图)所示。在亚油酸甲酯与LOX-1发生相互作用的过程中,有LEU541、HIS499和GLY701等24个氨基酸残基与亚油酸甲酯之间存在范德华相互作用力,氨基酸残基SER491、HIS494和ARG707与亚油酸甲酯之间存在碳氢键,氨基酸ARG707与其存在氢键作用。
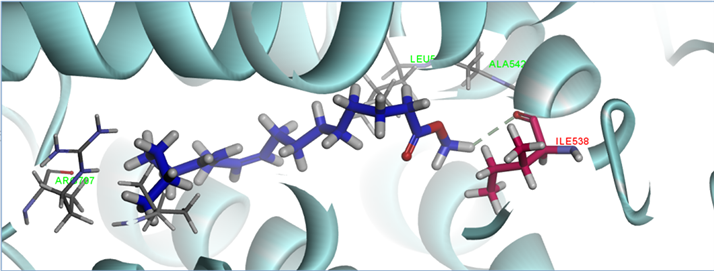 红色结构模型为脂氧合酶上的氨基酸残基,由红色字体标注出氨基酸残基名称;蓝绿色部分为脂氧合酶大分子模型;蓝色部分为亚油酸结构模型。
红色结构模型为脂氧合酶上的氨基酸残基,由红色字体标注出氨基酸残基名称;蓝绿色部分为脂氧合酶大分子模型;蓝色部分为亚油酸结构模型。
Figure 5. Structural model of soybean lipoxygenase-1 and methyl linoleate complex
图5. 大豆脂氧合酶LOX-1和亚油酸甲酯复合物的结构模型
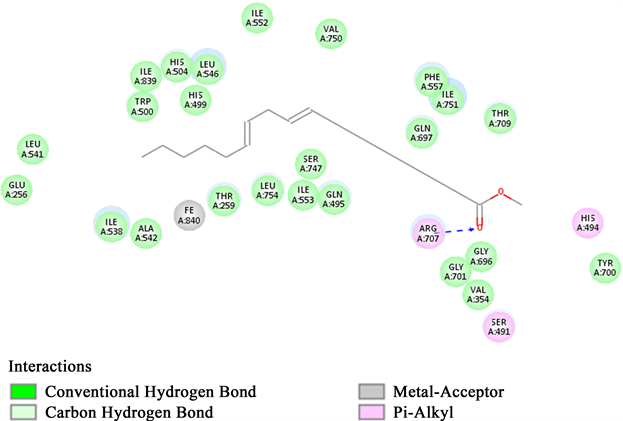
Figure 6. 2D docking diagram of soybean lipoxygenase-1 and methyl linoleate
图6. 大豆脂氧酶LOX-1和亚油酸甲酯的2D对接作用图
3.4. 对接结果分析
表1为亚油酸、亚油酸甲酯与亚油酸与LOX-1的对接结果,包括CDOCKER结合能、氢键的键长和键角。亚油酸与LOX-1分子形成的复合物具有的CDOCKER能为−37.60 kJ/mol,并在活性点位产生1个分子间氢键。通过调出Ligand Interactions功能,打开键的类型与键长显示,亚油酸上的O1作为电子供体与ARG707上的H原子形成的氢键键长为1.98 Å,键角为157.4˚。亚油酸甲酯与脂氧酶蛋白发生相互作用具有的CDOCKER能为−57.69 kJ/mol,有2个分子间氢键,亚油酸甲酯上的O2作为电子供体与ARG707上的H原子形成的氢键键长分别为1.85 Å和1.96 Å,键角分别为141.1˚和137.5˚。这些氢键作用力可以很好帮助底物小分子结合在脂氧酶蛋白上。

Table 1. Docking results of linoleic acid, methyl linoleate and soybean lipoxygenase LOX-1
表1. 亚油酸、亚油酸甲酯与大豆脂氧酶LOX-1对接结果
4. 结论
本实验在pH6和pH9的条件下考察了脂氧酶催化亚油酸、亚油酸甲酯的反应。氢过氧化亚油酸的产率在pH9和pH6条件下分别为58.5%和28.6%,而氢过氧化亚油酸甲酯的产率分别为14.2%和16.8%。运用Discovery Studio 2.5的CDOCKER分子对接模块研究LOX-1与亚油酸、亚油酸甲酯的对接情况,两者的CDOCKER结合能分别为−37.60 kJ/mol和−57.69 kJ/mol。说明LOX-1与亚油酸结合更稳定,反应活性也更高,与实际实验结果相符。此外,亚油酸及其甲酯与脂氧酶LOX-1作用过程产生氢键。
参考文献
NOTES
*通讯作者。