1. 引言
表观遗传是指不改变基因型而改变表型的基因表达改变的现象或机制。目前表观遗传学在基因调控对疾病影响方面提出了很多思路,并且肯定了一些疾病的发生受表观遗传学的影响,如过敏性鼻炎、过敏性哮喘等疾病的发生与表观遗传学存在着密切的联系 [1] 。DNA甲基化是一种被广泛研究的表观遗传修饰方式,与组蛋白修饰等方式一起,在调控基因表达和染色质构象等方面发挥了重要的作用。DNA甲基化功能是最早发现的基因表观修饰方式之一,即在DNA甲基化转移酶(DNMTs)的作用下使CpG二核苷酸5’-端的胞嘧啶转变为5’-甲基胞嘧啶,这一过程主要发生在CpG的胞嘧啶上 [2] 。DNA甲基化既可以调控转录水平,又可以在染色体结构稳定性、基因印记、X染色体失活等方面发挥作用 [3] 。因此DNA甲基化作为表观遗传学中实验研究较多、技术相对完善的一种方法具有很好的应用价值,同时也对深入理解基因表达、个体发育以及疾病的发生、发展机制都具有重要意义。
特禀体质是中医体质类型中的一种,它是由于先天禀赋不足和禀赋遗传等因素造成的一种特殊体质类型,其成因与先天禀赋不足、遗传因素、环境因素、药物因素等密切相关 [4] 。最近的研究已经发现特禀体质人群与过敏性疾病发生发展的关联性很高 [5] [6] [7],而由于地理环境、生活习惯和气候的独特性,内蒙古地区已成为特禀体质人群的集中区域,进而成为过敏性疾病的高发病地区 [8] [9] 。然而,对其在遗传水平表观的调节机制上,如通过其DNA甲基化的作用,我们知之甚少。在这项研究中,我们的目的是比较特禀体质(过敏组)和平和体质(正常组)的DNA甲基化图谱,以确定差异表达的基因,并进行了DNA甲基化水平和差异表达的基因之间的关联分析。
2. 材料与方法
2.1. 研究对象
本课题的研究对象均来源于2016~2017年内蒙古医科大学第一附属医院体检中心,通过前期进行的临床流行病学调查,共收集到2000余份体质量表,并以此为基础按照中医体质辨识结果及纳入、排除标准进行筛选,由经过培训的具有中医知识的研究生进行横断面现场调查,当场收回并确认资料合格。最终得到特禀体质154例及平和体质170例作为实验样本选择的范围人群。从范围人群中随机选择了4例研究对象进行全基因组DNA甲基化实验,其中特禀体质2例,平和体质2例。并从剩余范围人群中随机选择10例研究对象进行后续qPCR实验验证,其中特禀体质与平和体质人数各5例。两组受试对象性别均衡,年龄在20~60岁之间,无统计学差异。
2.1.1. 纳入标准及排除标准
纳入标准:
1) 符合特禀体质、平和体质判定标准者;
2) 祖辈三代均生活在内蒙古自治区境内;
3) 年龄满15周岁以上,80周岁以下;
4) 不患有传染性、精神类等影响试验的疾病;
5) 受试者及其一级亲属均愿意接受临床试验者;
6) 签署知情同意书者;
排除标准:
1) 有传染性疾病、先天缺陷或精神异常不能自主思考者;
2) 问卷填写不完整或因故未能完成问卷者;
3) 未签署知情同意书者。
2.1.2. 样本采集
采血前告知受试者实验目的、意义及相关注意事项,然后签署知情同意书。嘱受试者采血前一天禁酒和劳累,女性避开月经期,在空腹12小时后于清晨使用EDTA抗凝管采集肘静脉血8 ml,并充分颠倒使EDTA和血液混合,以防出现局部凝血的问题,保证在8小时内成功分离单个核细胞。
2.2. 实验材料
2.2.1. 实验试剂
主要试剂:生产厂家
红细胞裂解液:北京天根生化科技有限公司
TRIzol® Reagent:美国Invitrogen公司
氯仿:中国医药集团上海化学试剂公司
异丙醇:国药集团化学试剂有限公司
无水乙醇:国药集团化学试剂有限公司
RNase-free水:Qiagen (中国)生物技术公司
DEPC:美国Sigma公司
EB:上海博光生物科技有限公司
Certified Low-Range Ultra Agarose:美国Bio-Rad公司
TruSeqTM RNA Sample Prep Kit:美国Illumina公司
TruSeq PE Cluster Kit v3-cBot-HS:美国Illumina公司
TruSeq SBS Kit (300cycles):美国Illumina公司
Picogreen:美国Illumina公司
UltraPure Agarose:美国ABI-invitrogen公司
SuperScript III RT反转录Kit:美国ABI-invitrogen公司
Sybr qpcr mix:美国ABI-invitrogen公司
2.2.2. 实验仪器
主要仪器:生产厂家
微量移液器:德国Eppendorf
台式低速离心机himac CT6E:厦门名大科技有限公司
无菌超净工作台SW-CJ-2D:苏州净化设备有限公司
Bio-Gen PRO200精密匀浆器:相马科学仪器(北京)有限公司
NanoDrop2000c微量紫外可见分光光度计:美国THERMO公司
小型水平电泳槽Mini-Sub Cell Gt Cell:美国Bio-Rad公司
磁力架:美国Invitrogen公司
TBS380:美国Invitrogen公司
台式高速冷冻离心机:美国THERMO公司
核酸浓度测定仪:美国THERMO公司
qPCR仪:美国Applied biosystems公司
电泳仪:美国Bio-Rad公司
凝胶成像仪:美国Bio-Rad公司
2.3. 方法
2.3.1. DNA甲基化实验方法
登记各样本编号,白细胞分离,提取DNA,并进行质检。在通过测试后的样本中加入一定比例的阴性对照(lambda DNA),并使用Covaris S220将基因组DNA随机打断至200~300 bp。对碎片化的DNA片段进行末端修复,添加A尾,并附加具有全部甲基化胞嘧啶修饰的测试接头。随后进行Bisulfite处理(采用EZ DNA Methylation Gold Kit,Zymo Research),经过处理,未发生甲基化的C变成U,而甲基化的C保持不变。进行PCR扩增,得到最终的DNA文库。构建文库后,首先使用Qubit2.0进行初步定量,将文库稀释至1 ng/μl,然后使用Agilent2100检测文库的插入长度。在达到预期的结果后,使用Q-PCR来准确量化文库的有效浓度(有效文库浓度 > 2 nM),以保证文库质量。文库检测合格之后,根据有效浓度和目标数据量的要求合并不同的文库,进行Illumina HiSeq/MiSeq测序。测序的基本原理是边合成边测序(Sequencing by Synthesis)。将四种荧光标记的dNTP、DNA聚合酶和接头引物加入重新测序的流式细胞中进行扩增,当每个测序簇延伸互补链时,每个被荧光标记的dNTP就能释放出相对应的荧光,序列发生器捕获荧光信号,并使用计算机软件将光信号转换为测序峰,从而获得待测片段的序列信息 [10] 。
2.3.2. qPCR实验方法
登记各样本编号,白细胞分离,提取RNA,并进行质检。按照以下步骤建立反转录反应体系(RNA均取200 ng,补水到10 μl):RNA 10 μl、Oligo-dT 1 μl、Random 1 μl混匀后离心,65℃,五分钟,结束后置于冰上。在上述反应体系中加入下列反应液:dNTP (10 μM) 1 μl、0.1 M DTT 2 μl、5 × Buffer 4 μl、RT酶1 μl混匀后离心。置于42℃,水浴一小时。取出后置于85℃,灭活逆转录酶反应10分钟。反应结束后置于−20℃待用。每个样本分别用待检测基因和内参基因引物扩增,每个反应做3个重复,按照以下体系建立扩增体系(20 μl):cDNA 2 μl、qPCR mix 10 μl、primer F 1 μl、primer R 1 μl、ddH2O 6 μl于ABI 7900qPCR仪上,按照以下条件反应:95℃ 2分钟、94℃ 20 s、60℃ 20 s、72℃ 30 s共进行40个循环 [11] 。
2.3.3. 统计学方法
采用SPSS 20.0软件分析。计量资料采用
± s表示,对符合正态性检验和方差齐性检验采用单因素方差分析(One-way ANOVA),不符合采用非参数检验,以P < 0.05为差异有统计学意义。
3. 结果
3.1. DNA甲基化测序结果
在本研究中,设置2例平和组和特禀组2人进行原始数据的分析,4个样本分别产生了361、505、363、366、854、706、321、750、877、334、567、882个纯读数,且其定位读数分别占相应读数的84.6%、83.92%、81.90%、82.02% (表1)。

Table 1. Data from genome-wide bisulfite sequencing: 1 and 2 normal T1 and T2 allergic groups
表1. 全基因组重亚硫酸盐测序产生的数据:1、2正常组T1、T2过敏组
为了表征不同环境条件下样本间基因组甲基化水平的差异,我们进行了差异甲基化分析,两组共确定了47,150个DMR,在CG型甲基化中,低甲基化相较高甲基化为高。而在CHG型和CHH型甲基化中结果则恰恰相反,高甲基化较低甲基化为高。但无论发生哪种甲基化类型,其甲基化区域均主要位于内含子区且比值很高(图1)。
我们将基因分为特定的基因特征:启动子,外显子,内含子,5’UTR和3’UTR等。并且在这些功能区域进行MLs评估。正常组与过敏组CG型甲基化比较结果显示,5’UTR区域检测到最低的MLs,但在内含子和3’UTR区域内增加,而且3’UTR的ML较内含子高。正常组与过敏组CHG型和CHH型甲基化比较结果显示,两者之间具有相似的发展模式,在内含子区域检测到最低的MLs,而在外显子及3’UTR区域反而增加,以3’UTR的ML增加更为显著(图2)。

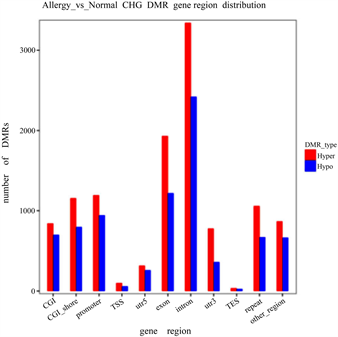

Figure 1. Display of DMR anchoring areas in three sequence environments (CG, CHG, CHH)
图1. 3种序列环境(CG, CHG, CHH) DMR锚定区域展示




Figure 2. Distribution of methylation levels on different genomic elements in samples from left one and two normal groups and right one and two allergic groups
图2. 样本甲基化水平在不同基因组元件上的分布左一和二为正常组,右一和二为过敏组
3.2. qPCR结果
通过对XKR6、PLCL1这两个甲基化位于内含子区的基因进行了qPCR实验验证,溶解曲线为单峰,引物特异性高,可以进行下一步定量实验验证(图3~5)。qPCR结果ct值用2−ΔΔCt检验,平和体质组表达为1.00,特禀体质组中XKR6基因结果为0.20,PLCL1基因结果为0.22,两个基因的表达均表现为下降(P < 0.05),(P < 0.01),差异有统计学意义(表2、图6)。

Table 2. Gene expression levels of endogenous constitution, and peace constitution (X ± sem)
表2. 特禀体质和平和体质两个基因mRNA表达量(X ± sem)
与平和体质组比较,*P < 0.05,**P < 0.01。


Figure 3. Gene actin amplification curve (left, middle) and dissolution curve (right)
图3. 基因actin扩增曲线(左、中)和溶解曲线(右)


Figure 4. Amplification curve (left, middle) and dissolution curve (right) of gene XKR6
图4. 基因XKR6扩增曲线(左、中)和溶解曲线(右)


Figure 5. Gene PLCL1 amplification curve (left, middle) and dissolution curve (right)
图5. 基因PLCL1扩增曲线(左、中)和溶解曲线(右)
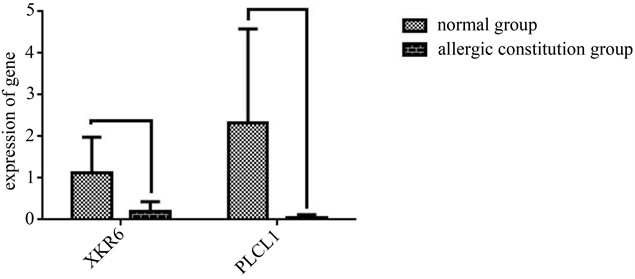
Figure 6. Two mRNA gene expression levels of constitution and peace and constitution
图6. 特禀体质和平和体质两个基因mRNA表达量
4. 讨论
DNA甲基化是表观遗传学研究的主要内容之一,近年来被发现与过敏性疾病发生密切相关。随着过敏性疾病的发病率日趋上升,全球约有30%~40%的人群受到该疾病不同程度的影响 [12],以哮喘为例,它与人类的基因及所暴露的环境有着非正常的交互关系,如肥胖、压力、暴露于空气污染、烟草污染环境等因素均会增加遗传易感性人群哮喘病的获得风险 [13],此外其引起的复发性呼吸困难导致全球每年约25万人的死亡 [14] 。基于表观遗传学机制将基因调控与环境线索及发展轨迹联系了起来,Avery DeVries [15] 等发现了哮喘早期表观预测因子的特征,并且指出了表观遗传学机制在调节本病的发生和易感性方面具有一定的潜在作用。有资料明确显示 [16] [17] 过敏性疾病的过敏体质有明显的遗传倾向性,且过敏性疾病是由于非基因序列改变所致的基因表达水平变化 [18] 。而表观遗传学可以较好的用来解释遗传与环境之间的关系,链接基因调控的环境线索和发展轨迹,并且在基因调控疾病影响方面提出了许多新思路,肯定了一些疾病的发生受表观遗传学的影响 [19] 。本次实验设置特禀质及平和质进行全基因组DNA甲基化测序,实验共获得47,150个差异甲基化读数,各种甲基化类型中,甲基化区域均主要位于内含子区且比值较高。表明在特禀质与平和质间基因甲基化存在明显差异,且研究前景广阔,运用DNA甲基化技术研究中医特禀体质,不仅填补了特禀体质相关研究的空白,丰富了其微观领域研究形式,还对其易感疾病的原因进行了探究,这为日后更好地改善体质,控制疾病发挥了重要的作用。
为了进一步验证特禀质组全基因组DNA甲基化测序结果与过敏性疾病发生关系,我们选取文献报道与过敏性疾病发病相关且在内含子区发生高倍甲基化的基因进行qPCR验证。与平和体质组比较,特禀体质组中XKR6,PLCL1相对表达均为下降(P < 0.05),(P < 0.01),差异有统计学意义。XKR6基因为Kell血型复合体亚基相关家族的成员6,位于染色体8p23位置上。目前国内学者对该基因的相关研究文献较少。国外学者在对嗜酸性粒细胞性食管炎(EoE)的研究中发现,EoE与过敏性病因学一致,常常与过敏性疾病共同发生,包括哮喘、湿疹以及食物过敏等 [20] [21] 。通过进一步研究还发现了另外两个新的基因组变异位点位于XKR6基因上,并且这几个全基因组易感性位点与EoE关系密切,在校正其特异性后效应大小没有显著影响 [22] 。在对自身免疫性疾病系统性红白狼疮(SLE)的研究中,ML Budarf [23] 等学者研究先前报道为SLE风险基因座的SNPs并观察了SLE12个不同位点上23个SNP与该病易感的相关性,结果显示包括XKR6、TNFSF4、IRF5在内的八个基因座位点的比值比高,同时研究人员还发现BLK和XKR6之间基因显著相关,虽然两个SNPs之间相距620 kb,但它们之间却存在较强的相关性。David [24] 等对53,862人进行了一项全基因组关联分析,包括自我报告的猫、尘螨、花粉等过敏。其使用广义估计方程来模拟过敏特异性遗传效应,鉴定了包括PLCL1中2q33.1在内的八个与哮喘有关的位点。结果表明,自我报告的过敏可以用来识别疾病易感性位点,并与过敏性致敏的研究一致,且发现了PLCL1等位点附近与自身免疫性疾病相关的新易感性位点,这一研究结果巩固并延伸了过敏性和自身免疫性疾病共有遗传病因学的证据。在一项包含美国、英国、欧洲等多地特应性皮肌炎(DM)患者的研究中,学者发现PLCL1不仅涉及以肌醇磷脂为基础的细胞内信号级联,还涉及受体的转换,因此它有助于维持肌肉紧张和γ-氨基丁酸介导的突触抑制。PLCL1可能与DM发病机制有密切关系,需要进一步研究 [25] 。qPCR验证结果显示出2个高甲基化的基因表达降低,这与高度DNA甲基化会抑制基因表达的论述相符,同时符合现阶段文献报道两个基因在过敏性疾病发生中的异常表达趋势。因此,在特禀体质和平和体质中内含子区域差异表达的基因可能是其易罹患过敏性疾病的关键,改变其基因的高甲基化状态可能为预防过敏性疾病提供了新的思路及方法。
过敏性疾病的发生因受基因及环境因素的共同影响,而表观遗传学研究为探讨过敏性疾病发生机制提供了新的思路,通过阐述表观遗传学改变与过敏性疾病发生之间的潜在关系将为疾病的诊断和治疗提供新的可能,并且突破了中医传统学术思想的束缚、拓宽了探索疾病发病机制及治疗手段的道路。与更具挑战性地组蛋白修饰分析不同,DNA甲基化作为一种蓬勃发展的技术更能代表表观遗传学在过敏性疾病研究中的总体。因此,本实验选用DNA甲基化技术研究中医特禀体质,不仅填补了特禀体质相关研究的空白,丰富了其微观领域研究形式,还对其易感疾病的原因进行了探究,这为日后更好地改善体质,控制疾病发挥了重要的作用。
基金项目
国家自然科学基金(No. 81460688)。
附录
附录1:知情同意书
知情同意书
尊敬的受试者:
您好!您将作为“基于表观遗传学对内蒙古特禀体质家族人群的基础性研究”课题的一名受试者,为了确保本次试验顺利进行并充分保障您的权益,在您同意参加之前,您需要清楚知道以下相关信息:
研究简介
我们正在进行一项临床调查工作,通过对具有家族遗传特点的特禀体质人群的免疫细胞即:CD4+Th2细胞的表观遗传改变进行研究来完善特禀体质的发病机理,探索一种新的思路和方法为临床防治特禀体质人群发生过敏性疾病提供理论依据和指导。
研究目的
研究具有家族遗传的特禀体质人群与平和体质人群Th2细胞在表观遗传学方面存在的差异,揭示家族遗传的特禀体质人群的表观遗传机理。将表观遗传学对特禀体质的影响理论应用到防治过敏性疾病方面。
调查过程概述
应用2009年版《中医体质分类与判定》标准筛选祖辈三代均生活在内蒙古地区的特禀体质(包含其一级亲属)和平和体质人群为研究对象,采集外周血进行DNA甲基化的实验研究。
获益与风险
通过本次调查能够较为客观地对您目前体质状况进行评价,并给出相应健康指导,有利于保持您的健康状态,同时有利于节省医疗成本及资源。您的参加将对中医体质理论的发展提供可贵的支持,期间可能会耽误您的宝贵时间,希望您能够支持我们的工作。
自愿参加原则
试验前请您对本次临床试验做详细的了解,我们会耐心解释您所关心的问题,然后由您自愿决定是否参与本次调查,参与与否都不会影响您和医生的关系及诊治。
资料保密
您的合法权益不会因为本项研究而受到侵犯,所有调查记录均属保密,姓名及相关信息不会出现在任何研究报告和公开出版物中。
受试者同意
调查人员已经把整个调查过程及潜在的风险与获益等内容完全向我解释清楚,我已提出自己的问题并得到了满意解答。
我作为受试者在此签名,表明我已清楚上述说明并同意参加该项调查。
受试者签名(正楷): 联系电话:
受试者家属签名(正楷): 调查医师签名(正楷):
日期: 年 月 日 日期: 年 月 日
附录2:伦理审批件

NOTES
*第一作者。
#通讯作者。